2. 山西省肿瘤医院 放射科, 太原 030000
2. Department of Radiology, Shanxi Province Cancer Hospital, Tainyuan 030000, China
肺结节作为肺癌早期诊断的重要依据,对其边缘精准分割显得格外重要。随着计算机断层扫描(Computed Tomography,CT)数据呈现出指数型增长的趋势,影像科医师面临着越来越繁重的CT图像审阅工作,即使医师能够快速准确地标记出结节所处的位置以及边界,但在长时间高强度的工作过程中难免会出现误诊漏诊。因此,急需一种新兴技术辅助医生诊断,而计算机辅助诊断(Computer Aided Diagnosis,CAD)[1-3]的出现给影像学带来了希望。辅助诊断技术的日趋成熟不仅能减少医生工作量,而且能够提高标记结节的准确率和效率。然而,由于肺部CT图像中结节的大小、形状以及其他临床特征(如钙化征、分叶征、毛刺征等)都存在明显差异,导致目前的一些分割方法的肺结节检出率低,耗时长。因此,构建一种高效的肺结节分割模型成为一个难点。
本文构建一种基于深度神经网络的肺结节分割模型以提高肺结节的检出率,减少肺结节检测时间,同时将双注意力模块融入深度神经网络,以优化小尺寸肺结节,进而提高多类型肺结节的分割精度。
1 相关工作近年来,深度学习的广泛使用使得研究人员开始利用神经网络提取肺结节的深层特征,从而对结节进行自动诊断,代替使用手工特征和描述符[4]的传统肺结节分割方法。文献[5]通过调整对比度以增强CT图像中肺结节部分,然后根据经验设定阈值与形态学操作的参数对图像进行预处理,最后利用简单的区域增长算法分割肺结节。文献[6]首先使用2D深度神经网络对肺部CT图像进行粗分割,然后利用概率图模型(马尔科夫模型)对粗分割结果进行优化以得到精确的分割结果。文献[7]提出一种多视图 2.5D卷积神经网络用于肺结节的分割,该网络由3个CNN分支组成,分别从一组结节的轴向视图、冠状视图和矢状视图中捕获敏感性特征,每个分支包括7个堆叠层,并以多尺度结节斑块为输入。3个CNN分支与1个全连接层相连,以预测斑块中心体素是否属于结节。文献[8]使用结节尺寸作为最主要的诊断标准,利用Mask R-CNN对肺结节进行分割进而得到轮廓信息。文献[9]提出将FCM算法作为基础,同时应用小波变换对CT图像展开分解,之后将分解后的低频图的像素点作为FCM算法的基础点,最后采用马氏距离进一步修正得到分割结果。但是上述方法均存在以下问题:
1)结节具有复杂的形状和高度异变性纹理,2D低层次描述符无法捕获辨识性特征。仅使用2D卷积神经网络提取的特征无法映射为高质量的分割特征图,从而影响网络训练的效率。CT图像本质上是三维数据,因此联系空间上下文信息对肺结节分割起着重要的作用。文献[6-7]分别使用2D、2.5D神经网络分割肺结节,但是单张的2D肺部CT图像不具备有效区分微小结节和血管剖面的能力,都没有充分利用肺结节的空间特征,导致分割精度较低。文献[8]利用结节的尺寸作为主要特征分割肺结节,但是却忽视了结节异变性的纹理特征和形状特征,进而无法对奇异性结节完整分割。
2)在相对小目标的分割问题中,建立局部特征与全局特征的相关性有助于提高特征表示,进而提高分割的精度。文献[6]虽然在分割网络的后端利用概率图模型提高分割精度,但是概率图模型只有得到较好的先验概率函数时,才能更精准地计算后验概率以优化第一阶段的分割结果,该方法无法自适应地根据结节的空间特征去分割。文献[5, 9]分别使用传统的区域增长算法和FCM算法作为分割方法的主框架,但都没有充分考虑肺结节局部特征与全局特征的相关性和依赖性,以至于对具有非规则形状特征的肺结节造成欠分割。
针对上述问题,本文提出基于3D-UNet网络的双注意力机制肺结节分割方法。UNet网络在医学图像分割领域有优秀的表现,为适应肺结节的分割,本文将原始的2D-UNet网络扩展为3D网络以捕获结节空间信息,并引入双注意力机制使网络的重点集中到关键特征区域以提高对小尺寸结节的分割精度。
2 DA 3D-UNet方法本文提出的DA 3D-UNet网络结构如图 1所示。首先,在3D-UNet网络的主框架中,本文使用最新提出的DUpsampling结构替代解码层路径中的传统上采样方法,恢复编码路径中结节的细节特征,提高结节特征图质量,加快网络收敛速度。其次,将双注意力模块,即空间注意力模块和通道注意力模块,应用于3D-UNet网络倒数第二层的特征图以捕获局部特征与全局特征的相关性及依赖关系,将网络注意力集中到病灶区域,进而提高分割精度。
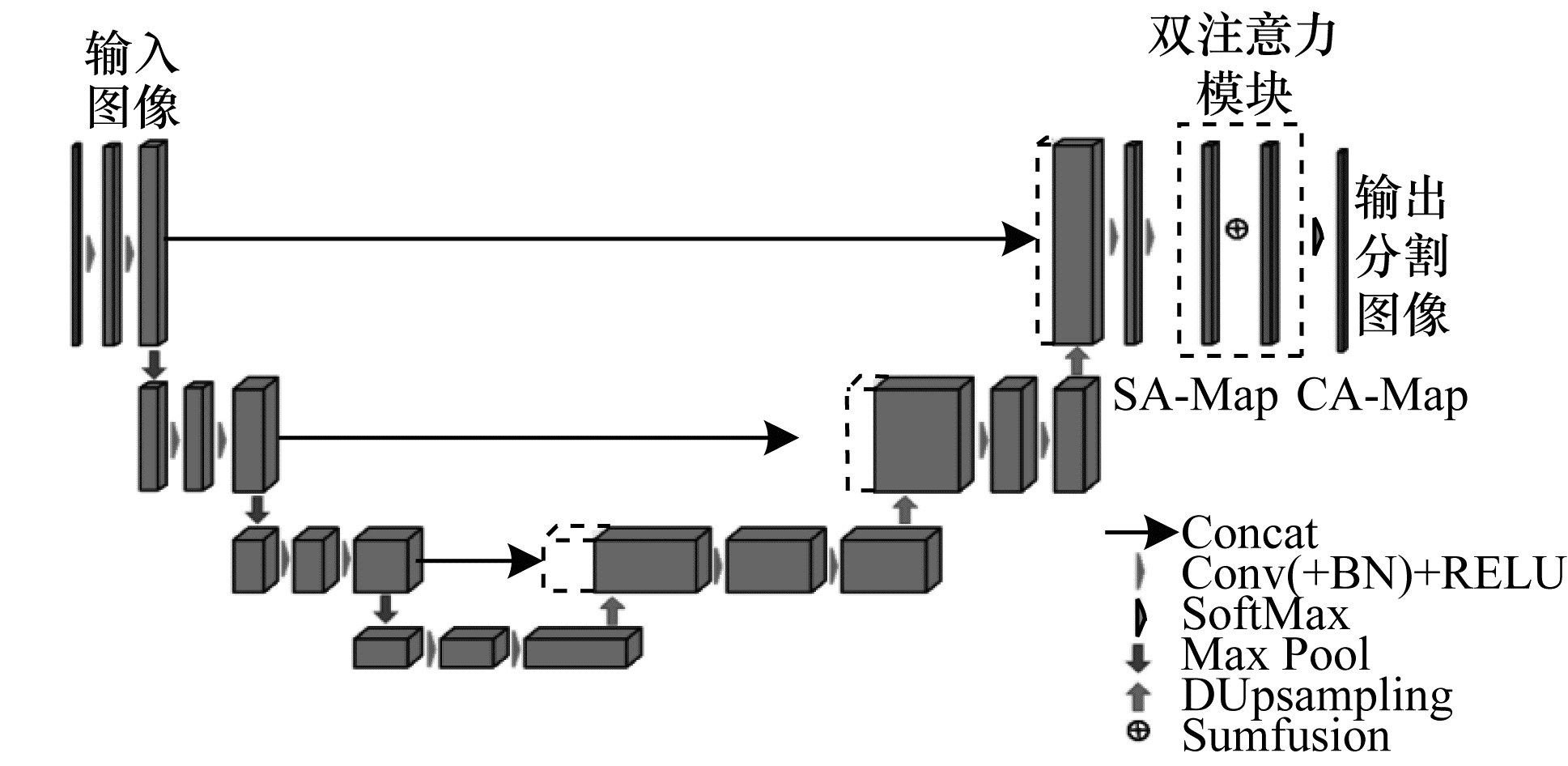
|
Download:
|
| 图 1 网络整体框架 Fig. 1 Network overall framework | |
DUpsampling结构是2019年TIAN[10]等人提出的一种基于数据相关性的新型上采样结构。上采样结构通常存在于分割网络的解码层中,其作用是将特征图恢复至原始图像的大小。基于双线性插值和最近邻插值的上采样操作虽然在一定程度上能够对卷积层提取的特征进行捕获和恢复,但是其过程没有考虑每个被预测像素之间的相关性,这种弱数据依赖的卷积解码器无法产生相对较高质量的特征图。本文将基于数据相关性的DUpsampling结构加入3D-UNet[11]网络重构编码路径[12]提取到的特征,使得到的特征图有更好的表达能力。在上采样的过程中,通过最小化特征图的像素点与被压缩标签图像之间的损失从而得到最“正确”的输出,具有很强的重建能力。DUpsampling的结构如图 2所示。
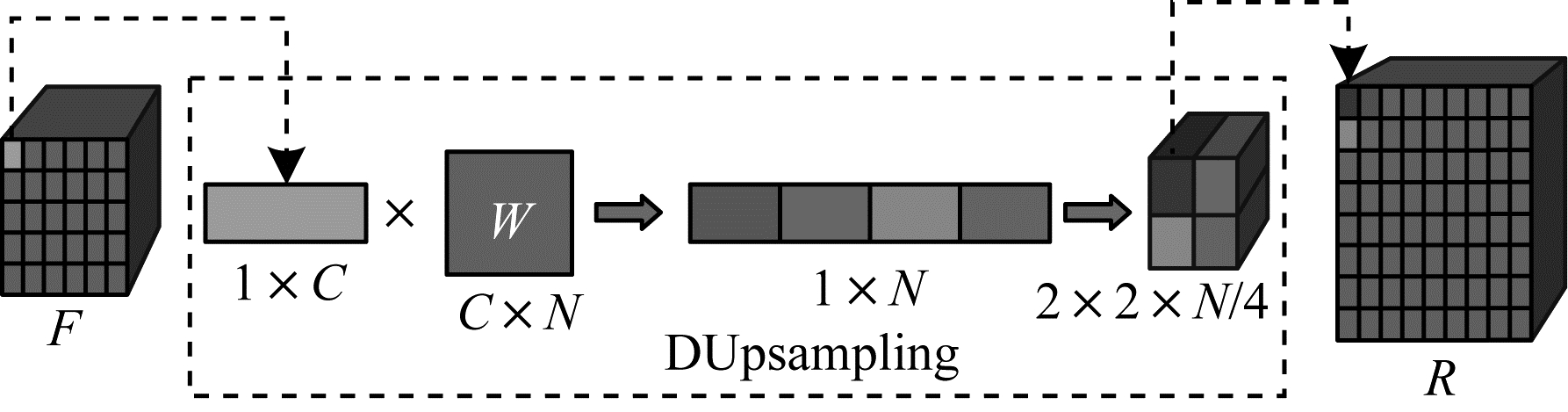
|
Download:
|
| 图 2 DUpsampling结构 Fig. 2 Architecture of DUpsampling | |
在图 2中,
| $ \begin{array}{l} \mathit{\boldsymbol{v}} = \mathit{\boldsymbol{x}} \times \mathit{\boldsymbol{W}}\\ \mathit{\boldsymbol{\tilde x = v}} \times \mathit{\boldsymbol{P}} \end{array} $ | (1) |
其中,矩阵P是矩阵W的反变换,
| $ \begin{array}{l} {\mathit{\boldsymbol{P}}^*}, {\mathit{\boldsymbol{W}}^*} = \mathop {\arg \min }\limits_{\mathit{\boldsymbol{P}}, \mathit{\boldsymbol{W}}} \sum\limits_\mathit{\boldsymbol{v}} {{{\left\| {\mathit{\boldsymbol{x}} - \mathit{\boldsymbol{\tilde x}}} \right\|}^2} = } \\ \mathop {\arg \min }\limits_{\mathit{\boldsymbol{P}}, \mathit{\boldsymbol{W}}} \sum\limits_\mathit{\boldsymbol{v}} {{{\left\| {\mathit{\boldsymbol{x}} - \mathit{\boldsymbol{xWP}}} \right\|}^2}} \end{array} $ | (2) |
传统的分割网络仅在最后的Softmax层计算预测结果与标签图像之间的损失,再通过反向传播更新权重优化网络。但DUpsampling结构在上采样部分就提前计算特征图与被压缩标签之间的损失,再通过网络整体的反向传播使解码层中低分辨率的特征图融入高层次语义特征,进而提高特征图的质量以便双注意模块挖掘空间信息与通道信息。
2.2 双注意力模块在双注意力模块中,本文首先使用不同膨胀率的空洞卷积操作[13]来捕获不同尺度的特征图信息,将包含多个尺度的结果特征图进行融合,对融合结果使用空间注意力模块和通道注意力模块。空间注意模块根据所有位置特征的加权和选择性地聚集每个位置的特征,使相似的特征相互关联。同时,通道注意力模块通过整合所有通道图之间的关联特征,选择性地强调相互依赖的通道特征图。最后将两个注意模块的输出相加,以进一步提高特征表示,进而有助于提高小尺寸结节分割精度。双注意力模块如图 3所示。
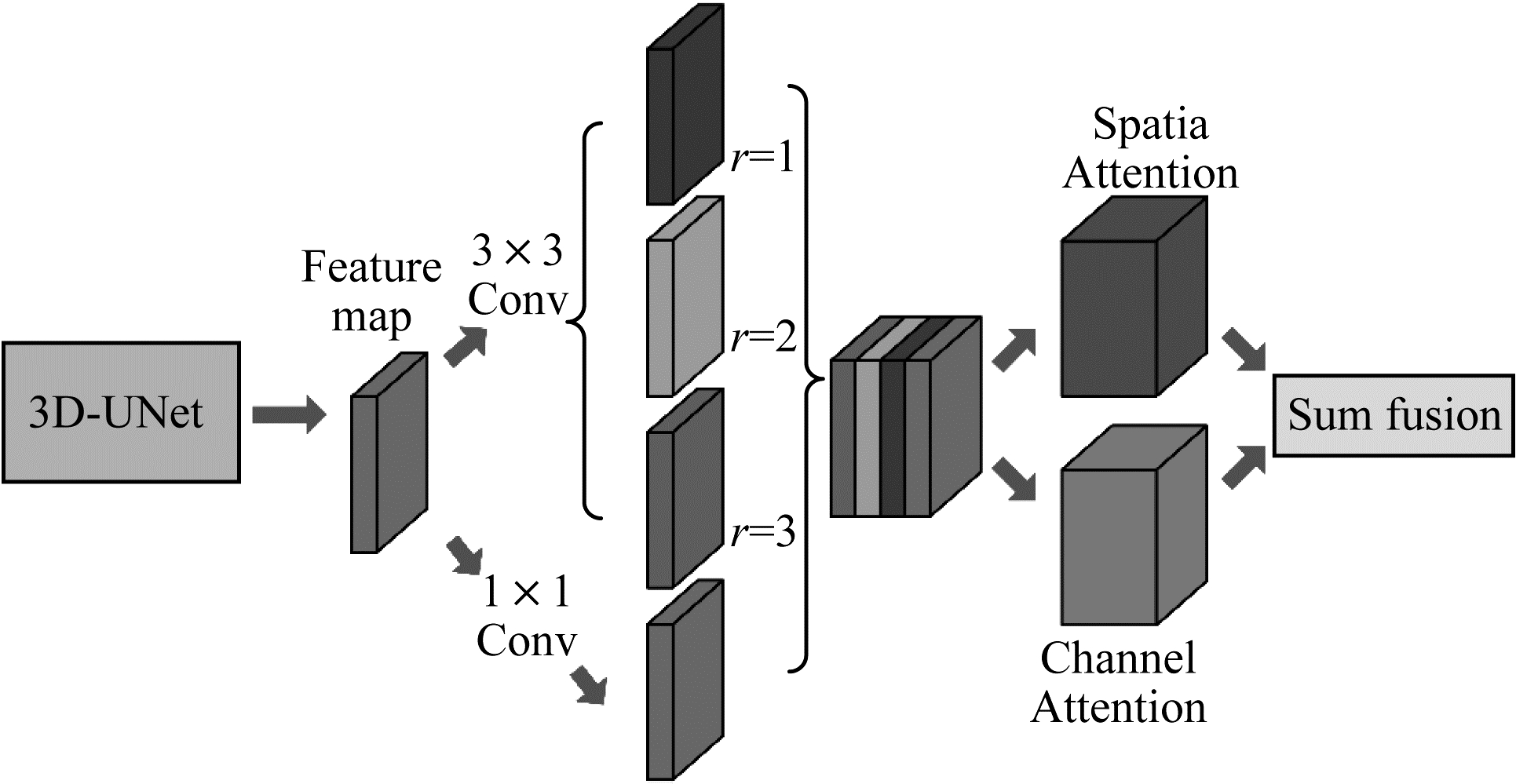
|
Download:
|
| 图 3 双注意力模块 Fig. 3 Double attention module | |
提取特征图的多尺度信息能够提高对小目标物体的分割精度。通常方法是将特征图经过多次最大池化操作得到不同分辨率的输出结果图再通过卷积层提取特征,但是经过多次池化操作会丢失小目标物体的细节信息甚至全部信息。肺结节在肺部CT图像占比很小,属于相对小目标类型分割。因此,本文引入不同膨胀率的空洞卷积对特征图进行特征提取,空洞卷积能够在不缩小特征图的情况下通过调整膨胀率以增大或缩小感受野,捕获多尺度特征图信息。
当给定输入特征图
| $ y\left( \mathit{\boldsymbol{x}} \right)\sum\limits_d {F\left( {\mathit{\boldsymbol{x}} + rd} \right)} \cdot \mathit{\boldsymbol{W}}\left( \mathit{\boldsymbol{x}} \right) $ | (3) |
其中,x是当前像素的位置,W是卷积核权重,r是膨胀率,d是当前卷积过程中的像素值。将标准形式的空洞卷积定义为Dconvr (F),其中Dconvr表示当膨胀率为r时对特征图F的空洞卷积操作。如图 3所示,在双注意力模块中将3D-UNet网络倒数第二层特征图作为输入,然后对该特征图执行级联空洞卷积操作,定义如下:
| $ \begin{array}{l} O\left( {C'} \right) = \\ \left\{ {{\rm{Dcon}}{{\rm{v}}_{{r_1}}}\left( F \right);{\rm{Dcon}}{{\rm{v}}_{{r_2}}}\left( F \right){\rm{Dcon}}{{\rm{v}}_{{r_3}}}\left( F \right);\mathit{\boldsymbol{M}}\left( F \right)} \right\} \end{array} $ | (4) |
其中,M代表对输入图像进行1×1的卷积得到的输出特征图,这里1×1的卷积操作是为了确保经过不同膨胀卷积的结果图之间通道保持一致,以融合不同尺度的肺结节特征。经过级联空洞卷积操作,最终得到一个融合多个尺度特征的特征图,该特征图将作为双注意力模块的输入。
2.2.2 空间注意力模块位置特征在分割任务中起重要的作用,它通过捕获像素间的上下文信息获得。由传统的特征提取网络而生成的局部特征未考虑临近像素的影响可能导致错误的分割。因此,为在局部特征上建立丰富的像素间位置关系,本文引入了空间注意力模块,如图 4所示。该模块通过将较大范围的上下文信息编码成局部特征,突出关键特征的位置,从而增强特征图表示能力。
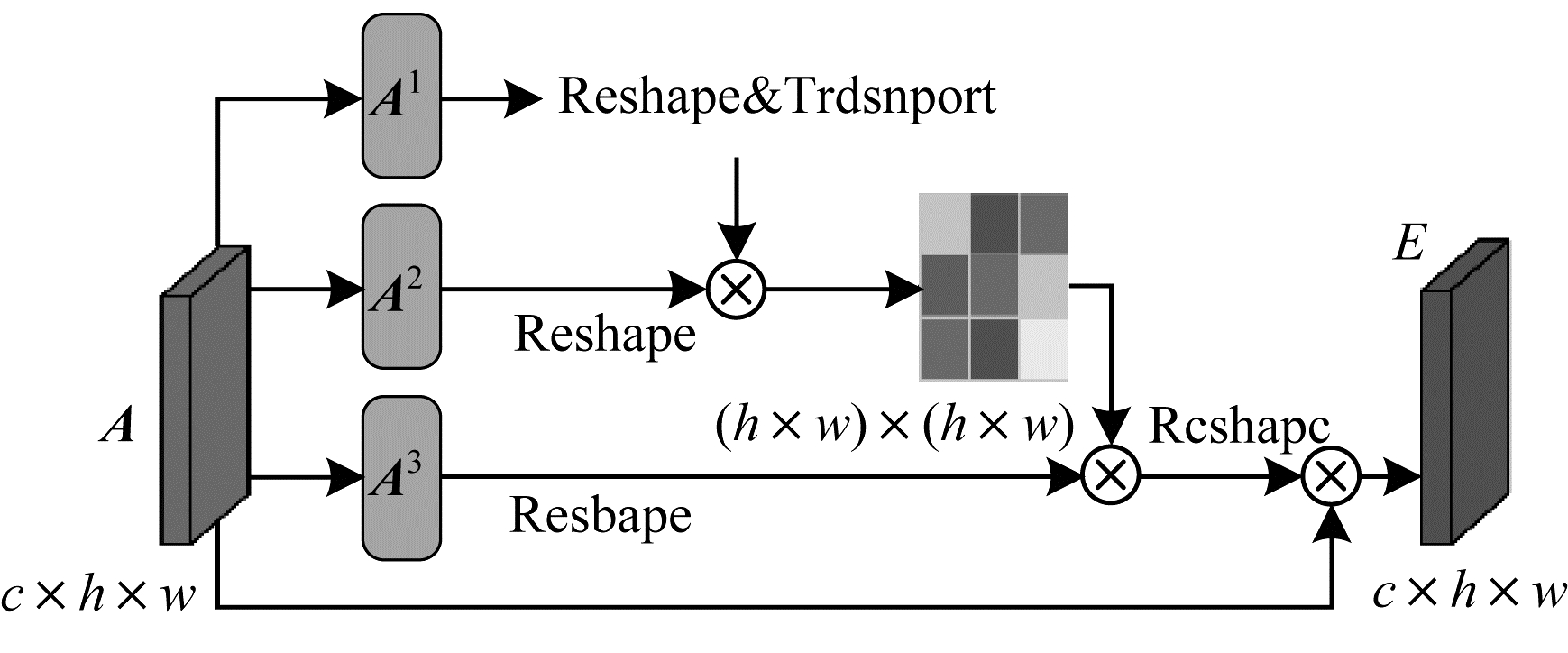
|
Download:
|
| 图 4 空间注意力模块 Fig. 4 Spatial attention module | |
如图 4所示,输入特征图A是融合不同膨胀率的空洞卷积结果的肺结节特征图,首先将其复制为3个新的特征映射,即特征图映射A1、A2、A3,且{A1, A2, A3}∈
| $ {s_{ji}} = \frac{{\exp \left( {\mathit{\boldsymbol{A}}_i^1 \cdot \mathit{\boldsymbol{A}}_j^2} \right)}}{{\sum\limits_{i = 1}^N {\exp \left( {\mathit{\boldsymbol{A}}_i^1 \cdot \mathit{\boldsymbol{A}}_j^2} \right)} }} $ | (5) |
其中,sji表示特征图中第i个像素位置对第j个像素位置特征的影响。两个位置的特征表示越相似,它们之间的相关性就越大,反之亦然。然后将重塑后的矩阵A3和矩阵S的转置进行矩阵乘法,并将结果重塑为
| $ {E_j} = \alpha \sum\limits_{i = 1}^C {\left( {{s_{ji}}\mathit{\boldsymbol{A}}_i^3} \right) + {\mathit{\boldsymbol{A}}_j}} $ | (6) |
其中,α初始化为0,并在训练过程中逐渐分配更多权重。从上述公式可知,在空间注意力图中每个位置的结果特征Ej是所有位置上的特征与原始特征的加权和。因此,它具有上下文信息并根据空间注意图有选择地聚合上下文,突出重点特征区域,提高分割精度。
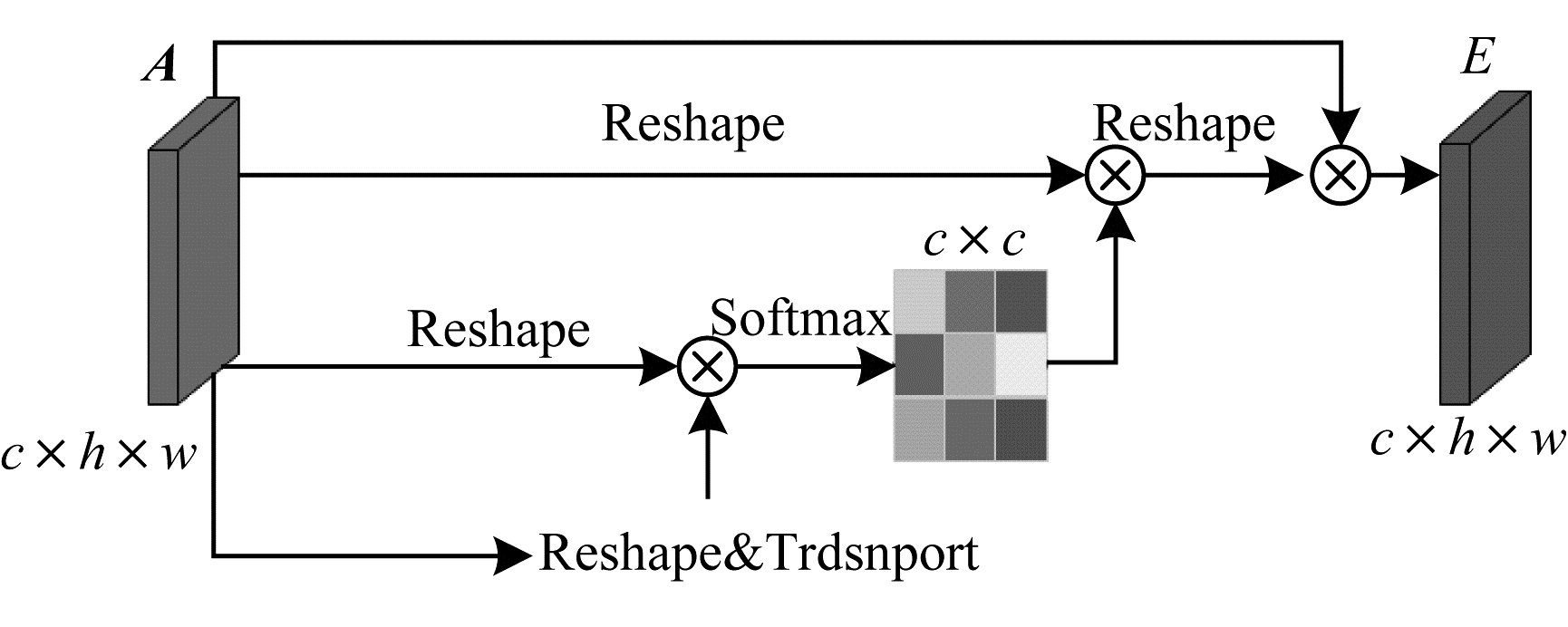
2.2.3 通道注意力模块高层特征的每个特征图的通道都可以看作是一个特定分割结果的响应,不同的语义响应相互关联。通过挖掘通道图之间的相互依赖关系,可以表现特征图的依赖关系,提高特定语义的特征表示。因此,本文构建了一个通道注意模块显式地建立通道之间的依赖关系,如图 5所示。
|
Download:
|
| 图 5 通道注意力模块 Fig. 5 Channel attention module | |
与空间注意力模块不同,通道注意力模块首先将特征图A重塑为
| $ {\mathit{\boldsymbol{x}}_{ji}} = \frac{{\exp \left( {{\mathit{\boldsymbol{A}}_i} \cdot {\mathit{\boldsymbol{A}}_j}} \right)}}{{\sum\limits_{i = 1}^C {\exp } \left( {{\mathit{\boldsymbol{A}}_i} \cdot {\mathit{\boldsymbol{A}}_j}} \right)}} $ | (7) |
其中,xji测量第i个通道对第j个通道的影响。此外,将x和矩阵A的转置矩阵进行矩阵乘法,并将其结果重塑为
| $ {E_j} = \beta \sum\limits_{i = 1}^C {\left( {{x_{ji}}{\mathit{\boldsymbol{A}}_i}} \right)} + {\mathit{\boldsymbol{A}}_j} $ | (8) |
其中,β初始化为0,并在训练过程中逐渐分配更多权重。每个通道的最终特征是所有通道特征和原始特征的加权和,从而建立了特征映射之间的长期语义依赖关系模型,它有助于提高特征的可辨别性,进而提高分割结果的完整度。
3 实验结果与分析 3.1 实验环境本文涉及的实验数据来自LIDC(Lung Imaging Database Consortium),排除了切片厚度大于2.5 mm的CT扫描图像,将剩余的888例肺部图像作为数据集,这888例CT图像中共包含1 186个结节,其直径范围为3.170 mm~27.442 mm。CT图像采集参数为150 mA、140 kV,平均层厚1.3 mm,图像分辨率为512像素×512像素。训练数据与测试数据分别为800例与88例。
在训练过程中,DA 3D-UNet以经过预处理后的10张连续CT图像为一组输入数据,使用MSRA[14]方法随机初始化权值,在标准反向传播更新中,学习速率初始化为0.1,每完成1个Epoch衰减5%,将批量大小设为64,动量设为0.9。使用10折交叉验证策略来评估该方法的性能,在训练和测试数据集中维持相近的数据分布情况,以避免由于数据不均衡而导致过分割和欠分割。
DA 3D-UNet网络搭建的环境为Python3.4,TensorFlow框架,CentOS7.4,NVIDIA GeForce1080Ti GPU,处理器IntelⓇ XeonTM CPU E5-2630 v4 @ 2.20 GHz。
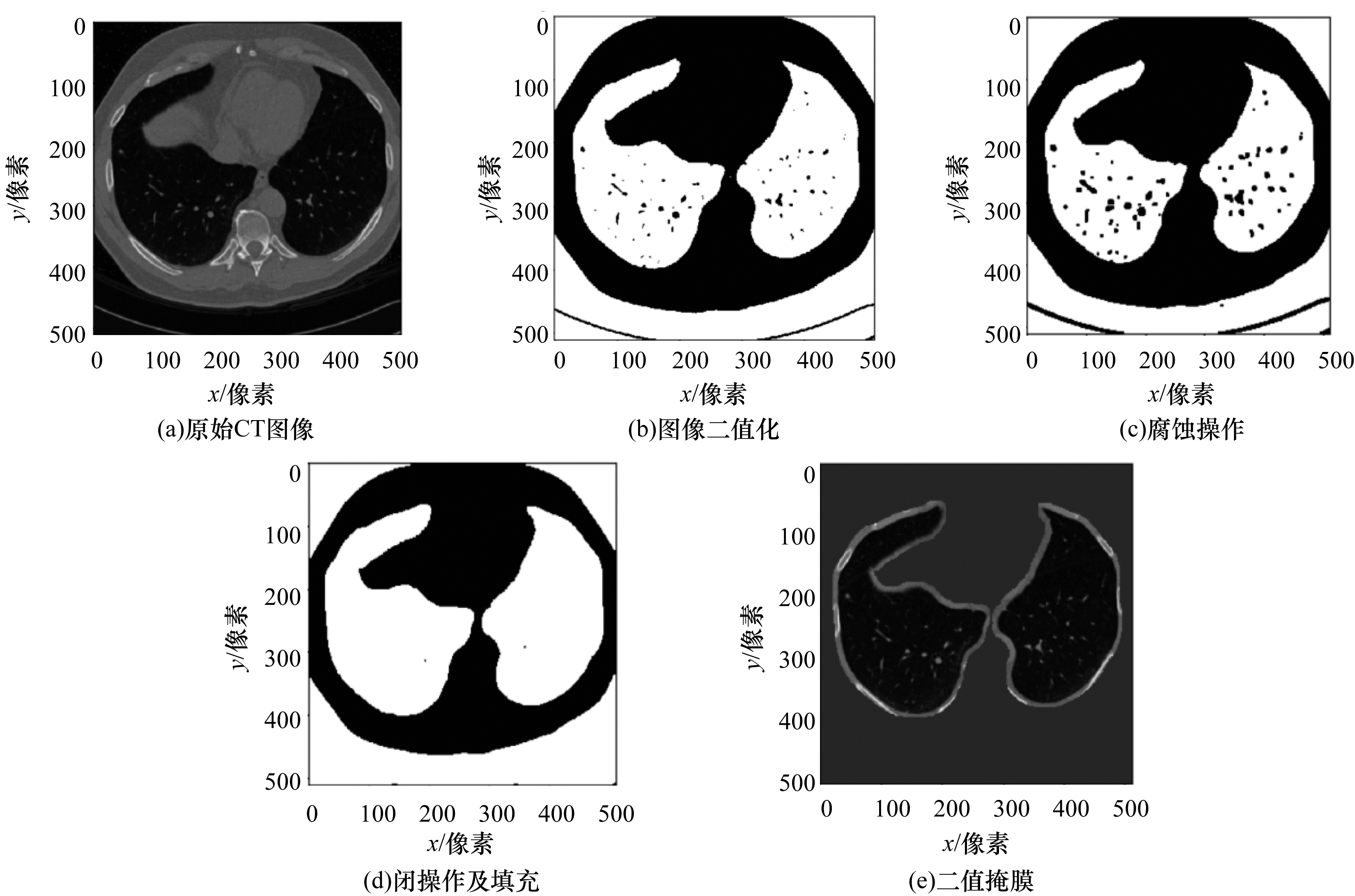
3.2 数据准备与评价标准 3.2.1 数据预处理本文提取左右肺叶区域掩模图作为模型输入,忽略胸腔及其他噪声部分,提取过程如图 6所示。
|
Download:
|
| 图 6 肺实质提取流程 Fig. 6 Extraction procedure of lung parenchyma | |
肺实质提取过程如下:1)二值化CT图像,通过聚类的方法找到可区分肺区域和非肺区域的阈值;2)Kmeans聚类,区分肺部区域是一类,非肺部周边为另一类;3)对图像中的高亮部分进行腐蚀操作,去除微小粒状噪声;4)进行膨胀操作,将血管侵吞为肺部组织,并且去除黑色噪声,特别是不透明射线造成的黑色肺部区域;5)将过程4)与原图进行数值型与操作并裁剪到相同大小得到肺实质区域。
3.2.2 数据扩充将每个CT标准化扫描平均值设置为-600,标准差为300,然后再进行数据扩充。数据扩充策略如下:
1)裁剪。对于每个512像素×512像素CT图像,每隔2个像素进行裁剪,裁剪为500×500的较小切片,因此每个候选区域的数据量增加36倍。
2)翻转。对于每个CT图像,从3个正交尺寸(冠状,矢状和轴向位置)进行翻转,因此最终为每个CT图像增加8×36=288倍的数据量。
3)重复。为平衡训练集中的正样本和负样本切片的数量,将正样本切片复制8次。
3.2.3 评价标准本文使用像素精度(Pixel Accuracy,PA)、平均像素精度(Mean Pixel Accuracy,MPA)和平均交并比(Mean Intersection over Union,MIoU)[15]3个国际通用的语义分割度量标准[16-18]来对分割结果做出评测。计算公式分别如式(9)~式(11)所示:
像素精度:
| $ {\rm{PA}} = \sum\limits_{i = 0}^k {{p_{ii}}} /\sum\limits_{i = 0}^k {\sum\limits_{j = 0}^k {{p_{ij}}} } $ | (9) |
平均像素精度:
| $ {\rm{MPA}} = \frac{1}{{k + 1}}\left( {{p_{ii}}/\sum\limits_{j = 0}^k {{p_{ij}}} } \right) $ | (10) |
平均交并比:
| $ {\rm{MIoU = }}\frac{1}{{k + 1}}\sum\limits_{i = 0}^k {\left( {{p_{ii}}/\sum\limits_{j = 0}^k {{p_{ij}} + \sum\limits_{j = 0}^k {\left( {{p_{ji}} - {p_{ii}}} \right)} } } \right)} $ | (11) |
肺结节的分割只需要得到一类语义分割结果(结节与背景),因此这里k=1。pij表示本属于i类却被预测为j类的像素数量。同理,pii与pji分别表示本属于i类被预测为i类的像素数量以及本属于j类却被预测为i类的像素数量。
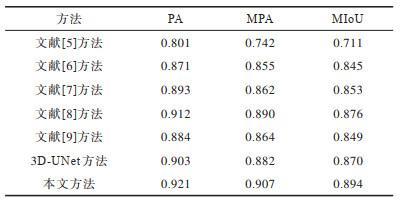
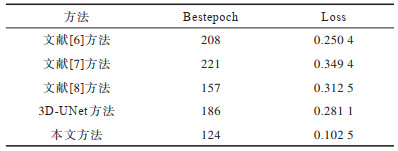
3.3 实验结果表 1是各种实验方法在88例测试数据上的实验对比结果。表 2是各种实验方法对88例测试数据集抽取的35例小尺寸结节(直径为3.170 mm~7.5 mm)的实验对比结果。表 3为神经网络迭代次数及损失(文献[5, 9]均不涉及神经网络),从表 3可以看出,本文方法在Bestepoch为124时损失值[19]就已经达到比较低的水平并且保持小幅度的浮动,其余方法的损失值均高于本文方法。
|
下载CSV 表 1 不同方法实验结果对比 Table 1 Comparison of experimental results of different methods |

|
下载CSV 表 2 小尺寸结节分割结果对比 Table 2 Comparison of small-size nodules segmentation results |
|
下载CSV 表 3 最优迭代次数及损失 Table 3 Optimal number of iterations and loss |
图 7为各类型肺结节的分割结果,其中,第1列、第2列是较为常见的孤立型肺结节,第3列、第4列为血管粘连型肺结节,第5列、第6列为胸膜牵拉型肺结节,第7列是较为少见的磨玻璃型肺结节,且第2、3、6列均为直径小于7.5 mm的小尺寸结节。本文提出的方法能够对大尺寸结节(第1、5、7列)进行完整的分割,对小尺寸结节(第2、3、6列)进行较为精准的分割,其余对比的方法或多或少都会存在过分割和欠分割[20]的情况。实验结果表明,本文提出的分割网络较优,在LIDC标准肺结节数据集[21]下肺结节分割的MIoU值达到89.4%。在图 7中第1行~第9行分别为CT图像、医师标注图像、文献[5]方法、文献[6]方法、文献[7]方法、文献[8]方法、文献[9]方法、3D-UNet方法和本文方法。

|
Download:
|
| 图 7 各类型肺结节分割结果 Fig. 7 Segmentation results of various-types lung nodules | |
针对目前分割网络存在的分割精度低及耗时长的问题,本文构建一种注意机制3D-UNet网络结构。将DUpsampling结构融入3D-UNet网络,在网络训练过程中提高上采样操作生成的特征图质量,使每次上采样后的特征图更加逼近标签数据,同时加快网络的收敛速度。在此基础上,提出空间注意力模块和通道注意力模块以分别捕获空间维度和通道维度上的全局依赖性。实验结果表明,该网络结构能够有效融合远程上下文信息,提高对大尺寸结节分割的完整度以及小尺寸结节的分割精度。下一步将分析各类型结节的特点,以实现全类型多变化结节的准确定位和追踪。
| [1] |
ZHANG Peng, XU Xinnan, WANG Hongwei, et al. Computer-aided lung cancer diagnosis approaches based on deep learning[J]. Journal of Computer-Aided Design & Computer Graphics, 2018, 30(1): 90. |
| [2] |
CHEN F, LI C, CABRERIZO M, et al.A novel Gaussian discriminant analysis-based computer aided diagnosis system for screening different stages of Alzheimer's disease[C]//Proceedings of the 17th IEEE International Conference on Bioinformatics and Bioengineering.Washington D.C., USA: IEEE Press, 2018: 325-336.
|
| [3] |
LBACHIR I A, ESSALHI R, DAOUDI I, et al.A new mammogram preprocessing method for computer-aided diagnosis systems[C]//Proceedings of AICCSAA'18.Hammamet, Tunisia: [s.n.], 2018: 584-596.
|
| [4] |
LOPEZ TORRES E, FIORINA E, PENNAZIO F, et al. Large scale validation of the M5L lung CAD on heterogeneous CT datasets[J]. Medical Physics, 2015, 42(4): 1477-1489. DOI:10.1118/1.4907970 |
| [5] |
EL-REGAILY S A, SALEM M A M, AZIZ M H A.Lung nodule segmentation and detection in computed tomography[C]//Proceedings of the 7th International Conference on Intelligent Computing and Information Systems.Washington D.C., USA: IEEE Press, 2017: 257-269.
|
| [6] |
MUKHERJEE S, HUANG X, BHAGALIA R R.Lung nodule segmentation using deep learned prior based graph cut[C]//Proceedings of 2017 IEEE International Symposium on Biomedical Imaging.Washington D.C., USA: IEEE Press, 2017: 178-199.
|
| [7] |
WANG S, ZHOU M, GEVAERT O, et al.A multi-view deep convolutional neural networks for lung nodule segmentation[C]//Proceedings of the 39th Annual International Conference of the IEEE Engineering in Medicine and Biology Society.Washington D.C., USA: IEEE Press, 2017: 2232-2245.
|
| [8] |
LIU Menglu, DONG Junyu, DONG Xinghui, et al.Segmentation of lung nodule in CT images based on mask R-CNN[C]//Proceedings of the 9th IEEE International Conference on Awareness Science and Technology.Washington D.C., USA: IEEE Press, 2018: 335-348.
|
| [9] |
LI Yue. Application of wavelet transform and improved FCM algorithm inmedical CT image segmentation[J]. Electronic Test, 2017, 23(22): 42-43. (in Chinese) 李越. 小波变换和改进的FCM算法在医学CT图像分割中的运用[J]. 电子测试, 2017, 23(22): 42-43. |
| [10] |
TIAN Zhi, HE Tong, SHEN Chunhua, et al.Decoders matter for semantic segmentation: data-dependent decoding enables flexible feature aggregation[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition.Washington D.C., USA: IEEE Press, 2019: 3126-3135.
|
| [11] |
CICEK O, ABDULKADIR A, LIENKAMP S S.3D-UNet: learning dense volumetric segmentation from sparse annotation[C]//Proceedings of International Conference on Medical Image Computing and Computer-Assisted Intervention.Berlin, Germany: Springer, 2016: 221-236.
|
| [12] |
VIJAY B, ALEX K, ROBERTO C. SegNet:a deep convolutional encoder-decoder architecture for scene segmentation[J]. IEEE Transactions on Pattern Analysis & Machine Intelligence, 2017, 39(12): 2481-2495. DOI:10.1109/TPAMI.2016.2644615 |
| [13] |
ZHANG Xiaohu, ZOU Yuexian, SHI Wei.Dilated convolution neural network with LeakyReLU for environmental sound classification[C]//Proceedings of the 22nd International Conference on Digital Signal Processing.Washington D.C., USA: IEEE Press, 2017: 202-215.
|
| [14] |
HE Kaiming, ZHANG Xiangyu, REN Shaoqing.Delving deep into rectifiers: surpassing human-level performance on ImageNet classification[C]//Proceedings of CVPR'15.[S.1.]: IEEE Computer Society, 2015: 109-118.
|
| [15] |
NOWOZIN S.Optimal decisions from probabilistic models: the intersection-over-union case[C]//Proceedings of IEEE Conference on Computer Vision & Pattern Recognition.[S.1.]: IEEE Computer Society, 2014: 548-555.
|
| [16] |
ZHOU Quan, YANG Wenbing, GAO Guangwei, et al.Multi-scale deep context convolutional neural networks for semantic segmentation[EB/OL].[2019-11-20].https://www.researchgate.net/publication.
|
| [17] |
BOUGET D, JORGENSEN A, KISS G, et al. Semantic segmentation and detection of mediastinal lymph nodes and anatomical structures in CT data for lung cancer staging[J]. International Journal of Computer Assisted Radiology and Surgery, 2019, 14(2): 1-10. DOI:10.1007/s11548-019-01948-8 |
| [18] |
LI S, AHMAD I, AVED A.A two layer pairwise framework to approximate superpixel-based higher-order conditional random field for semantic segmentation[C]//Proceedings of 2018 IEEE International Conference on Multimedia & Expo Workshops.Washington D.C., USA: IEEE Press, 2018: 557-569.
|
| [19] |
LI Pengcheng, YI Jinfeng, ZHOU Bowen.Improving the robustness of deep neural networks via adversarial training with triplet loss[C]//Proceedings of the 27th IEEE International Joint Conference on Artificial Intelligence.Washington D.C., USA: IEEE Press, 2019: 335-348.
|
| [20] |
RANZINI M B M, EBNER M, CARDOSO M J.Joint multimodal segmentation of clinical CT and MR from hip arthroplasty patients[C]//Proceedings of the 5th International Workshop and Challenge on Computational Methods and Clinical Applications in Musculoskeletal Imaging.Berlin, Germany: Springer, 2018: 431-446.
|
| [21] |
MCNITTGRAY M F, ARMATO S G, MEYER C R, et al. The lung image database consortium data collection process for nodule detection and annotation[J]. Academic Radiology, 2007, 14(12): 1464-1474. DOI:10.1016/j.acra.2007.07.021 |
 2021, Vol. 47
2021, Vol. 47
