2. 香港浸会大学 计算机科学系, 香港 999077;
3. 中电数据服务有限公司, 北京 100191;
4. 华北电力大学 控制与计算机工程学院, 河北 保定 071000
2. Department of Computer Science, Hong Kong Baptist University, Hong Kong 999077, China;
3. China Electric Data Service Co., Ltd., Beijing 100191, China;
4. School of Control and Computer Engineering, North China Electric Power University, Baoding, Hebei 071000, China
新型冠状病毒肺炎(Corona Virus Disease 2019,COVID-19)是由严重急性呼吸综合征冠状病毒(SARS-CoV-2)引起的疾病[1-2]。2019年,COVID-19疫情迅速蔓延至全球范围,对人类生命安全构成严重威胁。据约翰斯·霍普金斯大学发布的全球新冠肺炎疫情实时现状报告[3],截至2021年1月25日,全球新冠肺炎确诊病例累计超过9 925.7万例,死亡人数达213.1万,并且确诊/死亡人数呈现持续快速增长的趋势[4-5]。
SARS-CoV-2病毒具有高度传染性[6-7],其传播途径主要为直接传播、气溶胶传播和接触传播。直接传播是指患者通过咳嗽、打喷嚏和说话产生的飞沫在人群中近距离被吸入导致感染[8];气溶胶传播是指飞沫混合在空气中形成气溶胶,被他人吸入后导致感染;接触传播是指飞沫沉积在物体表面,而且由于SARS-CoV-2病毒在环境中极度稳定且隐蔽,可以在不同的物体表面附着数天[9],最终通过接触导致感染。相关研究结果表明,COVID-19的严重性不仅在于死亡率,更多取决于其在社区中传播速度快和感染率高的特点[10]。宾夕法尼亚州立大学研究人员指出,美国3月份的新冠病毒病例数量可能是最初报告的80倍,增长速度为最初报告的2倍。近日发表的一项研究通过对比SARS-CoV-2、SARS-CoV和中东呼吸综合征冠状病毒(MERS-CoV),指出SARS-CoV-2的致死率最低,仅高于流感,但是其传染性高于另外两类冠状病毒[11]。在致病性方面,结合临床观察可以发现,COVID-19感染患者典型的临床特征以发热、乏力、干咳为主要表现,同时,少数患者伴有鼻塞、流涕、腹泻等症状[12]。更严重的是,重症患者的病情会快速发展为急性呼吸窘迫综合征(ARDS)和多器官功能衰竭,甚至导致死亡。此外,还会出现白细胞或淋巴细胞计数减少等症状[13-15]。值得注意的是,基于目前的流行病学调查可知,SARS-CoV-2病毒的潜伏期通常为3天~14天,且有临床病例显示其潜伏期最长可达24天[16]。在病毒潜伏期间,患者无感染症状,潜伏期过后,患者可能出现类似于普通感冒的症状,甚至仍然表现为无症状感染者。综上所述,COVID-19具有高传染性、强致病性和长潜伏期等特点,对其进行精准、快速诊断存在极大的难度。
目前,核酸检测是确诊COVID-19最常见的方法[17-18]。该方法利用RNA逆转录和聚合酶链式反应相结合的技术(RT-PCR)检测病毒RNA片段,且只有通过核酸测试为阳性才能确诊。然而,RT-PCR筛选存在敏感性低的问题,即使可疑患者的RT-PCR结果为阴性,也无法完全排除患者被SARS-CoV-2感染的可能性[19-20]。此外,核酸测试存在耗时较长、需要专用测试盒等缺点,因此,需要进一步加快检测速度并降低成本。与此同时,据中国国家卫健委印发的《新型冠状病毒肺炎诊疗方案(试行第七版)》,影像学特征被列为新冠肺炎疑似病例三条临床特征之一,胸部影像临床诊断结果也可作为新冠肺炎病例诊断的标准[21]。文献[22]通过对计算机断层扫描(Computer Tomography,CT)和RT-PCR两种检测诊断方法进行对比实验分析指出,相较于RT-PCR方法,基于胸部CT图像的诊断更加快速高效。通常来说,COVID-19患者肺部具有典型的影像学特征,包括毛玻璃结节(Ground-Glass Opacities,GGO)、肺硬化、肺纤维化和多发性病变等[23-25]。
此外,相关研究也表明,影像学信息能够在COVID-19诊断中起到至关重要的作用[26-27]。但是,基于CT图像的人工分析和诊断过程对专业知识依赖性很高,对CT图像特征的分析比较耗时,早期难以观察到隐匿病变,且难以区分其他病毒性肺炎和细菌性肺炎。人工智能(Artificial Intelligence,AI)技术可将视觉影像信息转化为深层次特征信息,且这些信息是可量化的,有助于减少人工操作和提高精准定量分析的效率[28],其中,卷积神经网络(Convolutional Neural Network,CNN)在肺部疾病的诊断中已经能够达到非常优秀的性能[29-30]。因此,为避免当下过度依赖核酸检测进行COVID-19诊断的局限性,研究人员设计了多种基于深度学习的COVID-19检测诊断模型。这些模型不仅能够实现检测诊断方式的多元化,而且可使诊断过程更加快速高效,最关键的是,其准确率可以媲美核酸检测理想情况下高达95%以上的准确率[31]。最近,越来越多的研究人员开始关注COVID-19患者医学影像数据(X射线(X-ray)图像或CT图像)的获取,并尝试在此基础上构建基于深度学习的COVID-19检测诊断模型。
本文阐述基于深度学习的COVID-19检测诊断研究现状,对相关实验数据集和评价指标进行讨论和总结,介绍21种基于深度学习的COVID-19检测诊断模型,根据模型任务和影像数据类型划分为单任务/多任务检测诊断模型和基于CT/X-ray图像的检测诊断模型,并对具有代表性的模型从骨干网络、数据集、数据类型、性能表现、分类种类和开源情况6个不同维度进行分析。在此基础上,调研抗疫场景下表现出色的人工智能应用实例,并针对基于深度学习的COVID-19检测诊断技术,探讨其未来的发展趋势。
1 数据集数据集是设计和训练一个深度学习模型的必要条件。因此,构建基于深度学习的COVID-19诊断模型必须收集医学影像数据集。
COVID-19影像数据类型主要是X-ray图像和CT图像,其对应的样本种类包含COVID-19、病毒性肺炎(Viral Pneumonia,VP)、细菌性肺炎(Bacteria Pneumonia,BP)、其他肺炎和正常(normal)等。对常见的8个公开数据集进行整理,具体见表 1。
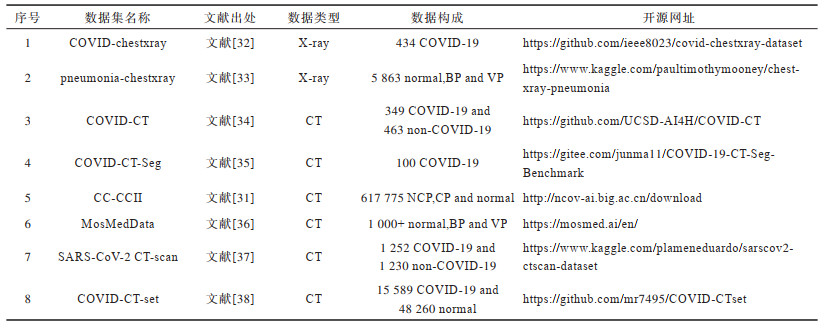
|
下载CSV 表 1 COVID-19相关的开源数据集 Table 1 Related open source datasets for COVID-19 |
常见的X-ray数据集主要包括COVID-chestxray[32]和pneumonia-chestxray[33],下文进行具体介绍。
1.1.1 COVID-chestxray数据集COVID-chestxray是一个公开的COVID-19病例数据集,包含COVID-19、其他病毒性肺炎和细菌性肺炎(MERS、SARS、ARDS)患者的胸部X-ray和CT图像,图像主要来自在线开源数据、网站和文献[39]中所提到参考文献。此外,数据集作者允许用户通过github网站向该数据集提交其他COVID-19数据。该数据集主要包含434张COVID-19的X-ray正面视图,只有少量的CT图像。
1.1.2 pneumonia-chestxray数据集pneumonia-chestxray数据集由训练集、测试集和验证集组成,包含健康受试者、非COVID-19病毒性肺炎患者和细菌性患者的5 863张X-ray图像。其中,前后胸部X-ray图像选自广州市妇幼保健中心的1岁~5岁儿童患者的回顾性研究。数据集作者对所有数据进行了筛查,通过去除所有低质量或不可读的扫描图来进行质量控制。然后,由两名专业医师对图像进行诊断并给出对应的标签。此外,为尽可能减少人为标记带来的误差,由第三位专家对前两位医师的标记结果进行检查。值得注意的是,pneumonia-chestxray是非COVID-19数据集,但是大量研究者构建的COVID-19数据集部分采用该数据集中的健康受试者、病毒性肺炎患者和细菌性肺炎患者的X-ray数据进行增强。
1.2 CT数据集常见的CT数据集主要包括COVID-CT[34]、COVID-CT-Seg[35]、CC-CCII[31]、MosMedData[36]、SARS-CoV-2 CT-scan[37]和COVID-CT-set[38],下文进行具体介绍。
1.2.1 COVID-CT数据集COVID-CT数据集包含216名COVID-19患者的349张CT切片和非COVID-19患者的463张CT切片。该数据集主要收集来自网站和出版物的医学影像,即从760份COVID-19相关预印本文献中收集,这些文献主要来自medRxiv和bioRxiv。此外,对于每个CT图像,数据集作者还收集了从论文中提取的meta信息,如患者年龄、性别、位置、病史、扫描时间、COVID-19的严重程度和放射学报告。该数据集有两个主要缺点:首先,许多CT图像中含有CT扫描机或医生产生的标记,这可能对深度学习技术产生负面影响;其次,每个病人只有少数几张CT图像而不是一个完整的3D扫描体,无法使用3D CNN来挖掘肺部深层次信息。
1.2.2 COVID-CT-Seg数据集COVID-CT-Seg数据集是一个公开可用的CT数据集。数据集作者对来自意大利医学和介入放射学学会网站上的JPG数据进行处理,其中包含40多个COVID-19患者的100张轴向CT图像。这些经由放射学专家分割的图像包含毛玻璃、硬化和胸腔积液三类标签。该数据集于2020年4月20日进行更新,增加了20个标记良好的扫描样本数据,包括左肺、右肺和病变感染区域的标记。新增数据的注释工作由三位经验丰富的放射科医生全程参与,其中两位放射科医生进行标记,一位进行验证。
1.2.3 CC-CCII数据集CC-CCII数据集是一个可公开使用的CT数据集,是目前针对COVID-19建立的最大CT数据集之一,包含4 154名患者的6 752次扫描结果,共计617 775张CT切片,包括新型冠状病毒性肺炎(Novel Corona Pneumonia,NCP)、普通肺炎(Common Pneumonia,CP)和正常对照组(normal)三大类。其中,肺炎包括细菌性肺炎和病毒性肺炎。然而,该数据集(2020年4月23日发布的1.0版)包含了一些错误,如某些扫描中存在CT图像次序混乱、部分扫描结果包含无用的头部信息但不包括关键肺部信息等。数据集作者于2020年7月3号发布v2.2版本,解决了压缩文件损坏问题,同时增加了对应的病变分割数据集。
1.2.4 MosMedData数据集MosMedData数据集包含1 000多位匿名患者的胸部CT扫描数据。数据集作者从2020年3月1日至4月25日在莫斯科通过统一放射信息服务(URIS)收集。此外,数据集中的所有CT数据均带有特殊标记,其根据分类进行标记以反映胸部CT肺组织中COVID-19的病理异常表现。数据集根据标记将CT数据分为五大类,即从CT-0(正常和无病毒性肺炎的CT)到CT-4(弥漫性玻璃膜混浊,肺实质受累超过75%)。同时,为了更好地开发AI算法,CT薄切片被转换为NIFTI格式。在相应的标记中,病例的整体标记用于患者的自动分类,定位标记用于为放射科医生指出在CT扫描中可疑的位置,病理轮廓标记用于肺部病变的自动定量评估和患者两次CT之间的动态评估。此外,专家明确标记了其中50个CT扫描集,每个CT切片上都标出了COVID-19特有的毛玻璃混浊和硬化的像素区域,并显示出肺组织异常。
1.2.5 SARS-CoV-2 CT-scan数据集SARS-CoV-2 CT-scan是一个公共可用的SARS-CoV-2 CT scan数据集,包含1 252例SARS-CoV-2感染阳性(COVID-19)的CT扫描数据和1 230例未感染SARS-CoV-2的患者的CT扫描数据。这些数据是从巴西圣保罗的医院的真实患者处收集的。该数据集发布的目的是鼓励研究和开发人工智能算法,旨在通过该数据集中的CT扫描数据进行训练测试分析,确定病人是否被SARS-CoV-2感染。
1.2.6 COVID-CT-set数据集COVID-CT-set数据集包含95例COVID-19患者的15 589张CT扫描图像和282例正常受试者的48 260张CT扫描图像,收集于伊朗的一家医疗中心。为了保护患者的隐私,数据集作者将数据格式从DICOM转换为TIFF。该数据集采用16位灰度数据,能够避免8位数据引起的信息丢失问题。此外,将图像的每个像素值除以其最大像素值,从而得到32位浮点类型的像素值,使得图像能够在常规的监视器中显示。
在X-ray数据集方面,pneumonia-chestxray包含大量非COVID-19肺炎数据,该数据集被广泛用于基于深度学习的其他肺炎检测诊断模型,是一个成熟且丰富的公共数据集,COVID-chestxray则由于包含COVID-19样本而被大多数研究人员所熟知。上述两个数据集通常以组合的形式,共同参与基于深度学习的COVID-19诊断模型的数据集构建。而CT数据集相对X-ray数据集拥有更多的COVID-19样本数据,甚至包含大量专家给出的标记(如MosMedData数据集),能够更好地训练基于深度学习的COVID-19检测诊断模型。但是,上述CT公开数据集提出时间较短,还未得到科研人员的广泛使用,更多的科研工作者使用私有数据集进行模型的训练。综上,数据集在数量和质量方面还有待进一步提高,使得科研人员构建的COVID-19检测诊断模型能够在一个公平完善的基础上进行比较。
2 评价指标基于深度学习的新冠肺炎诊断属于典型的医学图像处理问题,可以划分为分类任务和分割任务。对于分类任务,深度学习神经网络需要正确识别医学影像数据中的COVID-19和non-COVID-19,其中non-COVID-19可以进一步细分为病毒型肺炎、细菌型肺炎和正常等,根据分类的种类数量,新冠肺炎诊断可以是二分类问题,也可以是多分类问题。对分割任务,深度学习神经网络需要在对整个肺部进行分割预处理的基础上,进一步对肺部的病灶区域进行分割,从而获得医生最关注的区域,最终根据感染区域的特征和大小完成医学影像数据中新冠肺炎的诊断。下文分别对分类和分割任务中不同的评价指标进行介绍。
2.1 分类任务的评价指标分类任务涉及7种常用的评价指标,即表示敏感性的召回率(Recall)、精准率(Precision)、特异性(Specificity)、准确率(Accuracy)、F1-score、ROC(Receiver Operating Characteristic)曲线和AUC(Area Under Curve)指标[40-41]。
召回率、精准率和特异性指标分别如下所示:
| $ {R}_{\mathrm{R}\mathrm{e}\mathrm{c}\mathrm{a}\mathrm{l}\mathrm{l}}=\frac{{N}_{\mathrm{T}\mathrm{P}}}{{N}_{\mathrm{T}\mathrm{P}}+{N}_{\mathrm{F}\mathrm{N}}} $ | (1) |
| $ {P}_{\mathrm{P}\mathrm{r}\mathrm{e}\mathrm{c}\mathrm{i}\mathrm{s}\mathrm{i}\mathrm{o}\mathrm{n}}=\frac{{N}_{\mathrm{T}\mathrm{P}}}{{N}_{\mathrm{T}\mathrm{P}}+{N}_{\mathrm{F}\mathrm{P}}} $ | (2) |
| $ {S}_{\mathrm{S}\mathrm{p}\mathrm{e}\mathrm{c}\mathrm{i}\mathrm{f}\mathrm{i}\mathrm{c}\mathrm{i}\mathrm{t}\mathrm{y}}=\frac{{N}_{\mathrm{T}\mathrm{N}}}{{N}_{\mathrm{T}\mathrm{N}}+{N}_{\mathrm{F}\mathrm{P}}} $ | (3) |
其中,NTP表示实例为阳性并且被预测为阳性所对应的数量,NFN表示实例为阳性但被预测为阴性所对应的数量,NFP表示实例为阴性但被预测为阳性所对应的数量,NTN表示实例为阴性并且被预测为阴性所对应的数量。
由于召回率和精准率被作为单一指标进行评估,因此无法全面地评估诊断结果。同时,针对不同场景的分类应用,对召回率和精准率的要求也有所不同。因此,出现了F1-score。该指标综合考虑了以上两个指标,可以表示为两者的调和平均,如式(4)所示:
| $ {F}_{1}=\frac{2\cdot {P}_{\mathrm{P}\mathrm{r}\mathrm{e}\mathrm{c}\mathrm{i}\mathrm{s}\mathrm{i}\mathrm{o}\mathrm{n}}\cdot {R}_{\mathrm{R}\mathrm{e}\mathrm{c}\mathrm{a}\mathrm{l}\mathrm{l}}}{{P}_{\mathrm{P}\mathrm{r}\mathrm{e}\mathrm{c}\mathrm{i}\mathrm{s}\mathrm{i}\mathrm{o}\mathrm{n}}+{R}_{\mathrm{R}\mathrm{e}\mathrm{c}\mathrm{a}\mathrm{l}\mathrm{l}}} $ | (4) |
在更一般的情况下,该指标对应的形式如式(5)所示,其中,β为加权系数,用于调节召回率和精准率之间的权重关系。
| $ {F}_{\beta }=\frac{(1+{\beta }^{2})\cdot {P}_{\mathrm{P}\mathrm{r}\mathrm{e}\mathrm{c}\mathrm{i}\mathrm{s}\mathrm{i}\mathrm{o}\mathrm{n}}\cdot {R}_{\mathrm{R}\mathrm{e}\mathrm{c}\mathrm{a}\mathrm{l}\mathrm{l}}}{{\beta }^{2}\cdot {P}_{\mathrm{P}\mathrm{r}\mathrm{e}\mathrm{c}\mathrm{i}\mathrm{s}\mathrm{i}\mathrm{o}\mathrm{n}}+{R}_{\mathrm{R}\mathrm{e}\mathrm{c}\mathrm{a}\mathrm{l}\mathrm{l}}} $ | (5) |
F0.5-score和F2-score在统计学中均有广泛应用。新冠肺炎具有严重的危害性,在COVID-19诊断中必须保证足够高的敏感性,因此,推荐使用F2-score。
准确率表达式如下:
| $ {A}_{\mathrm{A}\mathrm{c}\mathrm{c}\mathrm{u}\mathrm{r}\mathrm{a}\mathrm{c}\mathrm{y}}=\frac{1}{N}({N}_{\mathrm{T}\mathrm{P}}+{N}_{\mathrm{T}\mathrm{N}}) $ | (6) |
其中,N表示样本实例的总数量。
ROC曲线的横轴是伪阳性率(False Postive Rate,FPR),满足FPR=1-SSpecificity,纵轴是真阳性率(True Postive Rate,TPR),满足TPR=RRecall。具体地,给定一个阈值,对所有实例进行分类预测,从而计算得出对应的一组(FPR,TPR)。最理想的情况为TPR=1,FPR=0。因此,ROC曲线越靠近坐标(0,1),说明算法性能越优秀。此外,ROC曲线具备一个很好的特性,即当测试集中的正负样本分布发生变化时,ROC曲线能够保持不变。通常实际数据集中存在类不平衡现象,即正、负样本数量差异较大。因此,ROC曲线得到了广泛的应用。AUC表示ROC曲线下的面积,用以评估分类器的优劣,对应的数值越大,代表算法性能越好。
2.2 分割任务的评价指标分割任务涉及5种常用的评估指标,包括DICE系数、体积重叠误差(Volumetric Overlap Error,VOE)、相对体积差(Relative Volume Difference,RVD)、对称位置的平均表面距离(Average Symmetric Surface Distance,ASD)和对称位置的最大表面距离(Maximum Symmetric Surface Distance,MSD)。
首先定义标识符Vgt和Vseg,以Vgt表示ground truth对应的分割结果,以Vseg表示预测的分割结果。
DICE系数作为分割任务中最常用的评价指标被广泛使用,其表达式如下:
| $ {D}_{\mathrm{D}\mathrm{I}\mathrm{C}\mathrm{E}}=\frac{2\cdot ({V}_{\mathrm{s}\mathrm{e}\mathrm{g}}\bigcap {V}_{\mathrm{g}\mathrm{t}})}{{V}_{\mathrm{s}\mathrm{e}\mathrm{g}}+{V}_{\mathrm{g}\mathrm{t}}} $ | (7) |
与DICE系数类似,VOE将交集运算替换为差集运算表示错误率,其表达式如下:
| $ {V}_{\mathrm{V}\mathrm{O}\mathrm{E}}=\frac{2\cdot ({V}_{\mathrm{s}\mathrm{e}\mathrm{g}}-{V}_{\mathrm{g}\mathrm{t}})}{{V}_{\mathrm{s}\mathrm{e}\mathrm{g}}+{V}_{\mathrm{g}\mathrm{t}}} $ | (8) |
RVD表示Vgt和Vseg两者体积间差异,其表达式如下:
| $ {R}_{\mathrm{R}\mathrm{V}\mathrm{D}}=\left(\frac{{V}_{\mathrm{s}\mathrm{e}\mathrm{g}}}{{V}_{\mathrm{g}\mathrm{t}}}-\mathrm{ }1\right)\times 100\mathrm{\%} $ | (9) |
为计算ASD,先定义Aseg表示预测的分割结果Vseg中的边界像素,Agt表示ground truth中的边界像素。由此计算Bseg:
| $ {B}_{\mathrm{s}\mathrm{e}\mathrm{g}}=\{\forall {p}_{1}\in {A}_{\mathrm{s}\mathrm{e}\mathrm{g}}, d({p}_{1}, {p}_{2})|\exists {p}_{2}\in {A}_{\mathrm{g}\mathrm{t}}\} $ | (10) |
其中,d表示边界像素之间的最短距离。同理可计算得到Bgt。根据计算得到的Bseg和Bgt,对应ASD的表达式如下:
| $ {A}_{\mathrm{A}\mathrm{S}\mathrm{D}}=\mathrm{m}\mathrm{i}\mathrm{n}\left(\right\{{B}_{\mathrm{s}\mathrm{e}\mathrm{g}}, {B}_{\mathrm{g}\mathrm{t}}\left\}\right) $ | (11) |
MSD则与ASD的定义类似,将其中的平均运算替换为最大值运算,其表达式如下:
| $ {M}_{\mathrm{M}\mathrm{S}\mathrm{D}}=\mathrm{m}\mathrm{i}\mathrm{n}\left(\right\{{B}_{\mathrm{s}\mathrm{e}\mathrm{g}}, {B}_{\mathrm{g}\mathrm{t}}\left\}\right) $ | (12) |
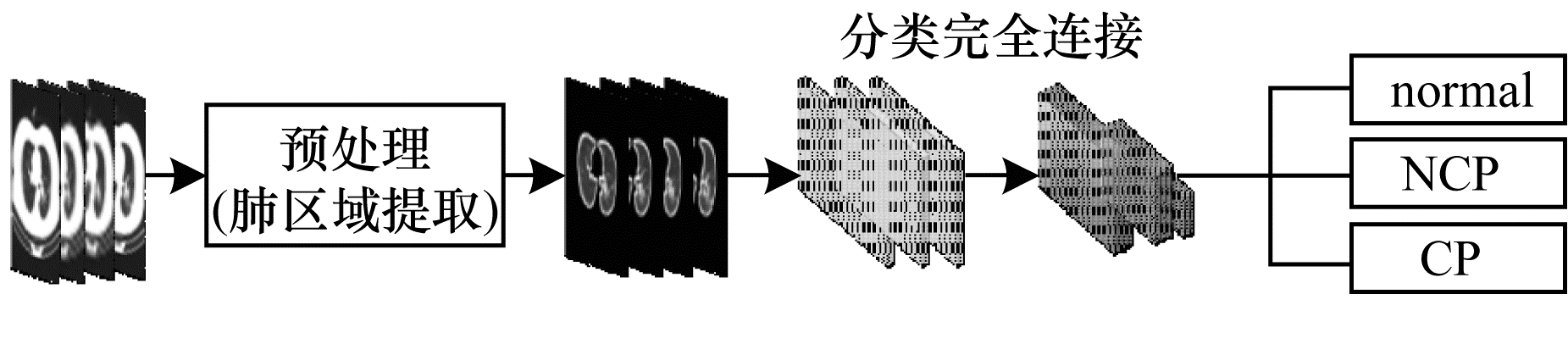
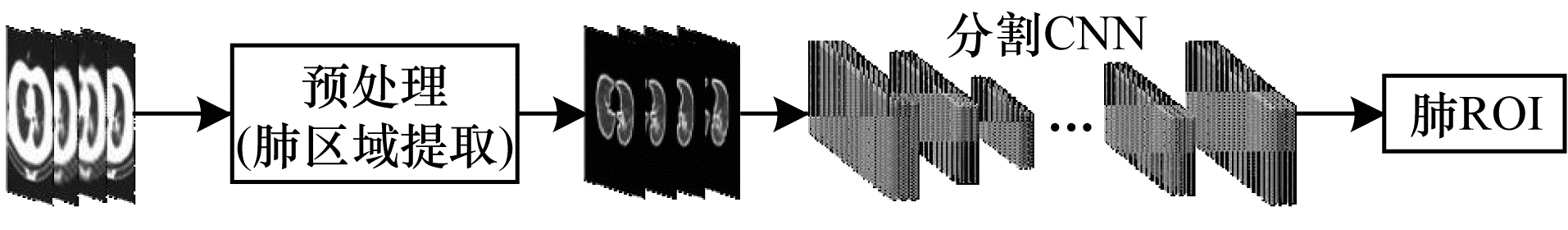
分别从模型任务(单任务即分类任务或分割任务,多任务即分类任务和分割任务)和影像数据类型(CT、X-ray)两个不同角度,对基于深度学习的COVID-19检测诊断模型进行分类与阐述。分类任务先对整个肺部区域进行提取,再使用卷积神经网络学习新冠肺炎的特征,从而实现对新冠肺炎的诊断;分割任务则在预处理(提取整个肺部区域)的基础上,进一步对肺部病灶区域进行了分割,能够更有效地对特征进行提取,从而改善新冠肺炎诊断的效果。但是,分割任务的实现依赖于额外的医学影像病灶标记,从现有数据集来看,相对于不带病灶标记的数据,带病灶标记的数据显得更加匮乏。基于深度学习的分类任务和分割任务处理流程分别如图 1和图 2所示,对两者进行组合即可得到多任务的处理流程。
|
Download:
|
| 图 1 基于深度学习的分类任务处理流程 Fig. 1 Processing flow of classification task based on deep learning | |
|
Download:
|
| 图 2 基于深度学习的分割任务处理流程 Fig. 2 Processing flow of segmentation task based on deep learning | |
文献[42]设计了一种深度学习模型,从典型的病毒性肺炎中筛选COVID-19。首先,放射科医生将感染区域标注为感兴趣区域(Region of Interest,ROI)。然后,作者构建基于Inception网络[43]的迁移学习神经网络用以提取特征向量,结合决策树和AdaBoost[44]对COVID-19和典型病毒性肺炎进行分类。该工作收集的数据集包括99例患者(44例COVID-19和55例典型病毒性肺炎)的胸部CT图像。该模型在内部测试数据集上的准确率为82.9%,特异性为84%,敏感性为81%。
文献[45]使用ResNet50[46]提取特征,并添加特征金字塔网络(Feature Pyramid Network,FPN)[47]和注意力模块[48]构建诊断模型。首先,提取主要肺部区域,并用肺本身填充肺分割的空白,以避免因不同肺轮廓引起的噪声。然后,设计细节关系提取神经网络(DRE-Net)来提取CT图像中的细节区域,并进行图像级别的预测。最后,将图像级别的预测集合相结合以实现患者级别的诊断。该系统对88例COVID-19患者、101例细菌性肺炎患者和86例健康受试者的CT扫描数据进行了深度学习模型的训练和验证。在测试集中,模型的AUC为0.95,敏感性为96%,其对肺炎分类(COVID-19或细菌性肺炎)的准确率为86.0%,对肺炎诊断(COVID-19或健康)的准确率为94.0%。
文献[49]使用了一个大型胸部CT数据集,包含3 322名患者的4 356张胸部CT图像(1 296张COVID-19、1 735张社区获得性肺炎(Community-Acquired Pneumonia,CAP)和1 325张非肺炎)。具体地,该方法将ResNet50模型应用于具有共享权重的二维(2D)切片上,结合最大值池化(max-pooling)以区分COVID-19、CAP和非肺炎。实验结果表明,该模型可以准确检测COVID-19,并能有效区分社区获得性肺炎和其他肺部疾病。该模型的敏感性为90%,特异性为96%,AUC为0.96。
文献[50]使用UNet++[51-52]提取COVID-19患者CT图像中的肺部病变区域,图像级别的标记(COVID-19或非COVID-19)直接根据分割的病变区域确定,而病例级别的标记则增加了一个连续图像预测结果的逻辑:将分割图像的预测结果分为四等份,只有当三张连续图像的病变分割出现在同一象限时,才判定病例为阳性。获得的数据集包括106例患者(51例为COVID-19,55例为其他疾病)的二维CT图像,且被随机分为训练集和验证集。该模型对COVID-19病例级别的检测诊断准确率达到95.2%,敏感性达到100%,特异性达到93.6%。目前该模型已被部署到武汉大学人民医院以帮助放射科医生进行新病例的分析,并在互联网上开源,以实现对CT图像的快速审查。此外,此项研究使用了包括27名潜在患者数据的前瞻性数据集做进一步验证,表明模型取得了与专业放射科医生相当的性能,可使放射科医生的阅读时间减少65%。
文献[53]提出一种基于DenseNet201的深度迁徙学习方法检测COVID-19,以判断患者是否被感染。该模型利用其在ImageNet数据集上学习的权值和卷积神经结构来提取CT图像中的特征。该文作者通过实验比较VGG16、Inception ResNet、ResNet152 v2和DenseNet201等模型的性能表现。实验结果表明,DenseNet201模型的F1-score和准确率均为最佳。
3.1.2 基于分割和分类任务的COVID-19诊断模型文献[54]使用ResNet18[55]自动检测CT图像中的感染区域,并使用ResNet23来识别感染区域是否为COVID-19症状。首先,利用ResNet18的分割模型从肺部CT图像中分割出候选感染区域。然后,利用ResNet23分类模型将候选区域分类到COVID-19、甲型流感病毒性肺炎或健康组,同时通过附带位置-注意力模型得到对应的置信分数。最后,利用噪声函数或贝叶斯函数计算感染类型和总置信度。CT样本数据集包括219例COVID-19患者、224例甲型流感病毒性肺炎和175例健康CT样本,该文作者随机抽取85.4%的CT样本作为训练集,其余14.6%的CT样本用于测试。在测试集上,该模型的敏感性达到了86.7%,相对不使用位置-注意力模型提高了3.4%。
文献[56]构造了一种用于筛选COVID-19患者的模型,该模型接收胸部CT图像,输出肺部异常定位和定量测量结果。模型主要由AB两个子系统构成。子系统A使用现成的商业软件对带有结节和病灶混浊的病例进行三维(3D)分析,子系统B对病例的切片进行二维分析,并使用U-Net分割网络提取感兴趣区域,且由2D ResNet50模型[57]检测出COVID-19相关异常结果,最终实现较大弥漫性混浊的定位和病例的诊断。模型根据阳性率(阳性检出切片与肺切片的百分比)确定病例分类。研究结果表明,该模型在以阳性率1.9%作为阈值的情况下,AUC为0.996,敏感性为96.4%,特异性为98%。
文献[58]收集了来自5家医院的1 136例患者(723例COVID-19阳性、413例COVID-19阴性)的胸部CT图像。该文提出的诊断模型包含基于UNet++的分割模型和基于ResNet50的分类模型。分割模型用于突出肺部区域所有病变,以便快速检查,同时作为分类模型的输入。该模型的敏感性和特异性分别为97.4%和92.2%,满足临床应用需求。
文献[59]提出一种多任务学习(Multi-Task Learning,MTL)模型检测COVID-19,并对胸部CT图像中的病变区域进行分割。MTL[60]将来自不同任务的若干信息进行组合,以提高模型的性能,使其具有更强的泛化能力。该模型由一个编码器和两个解码器组成,用于图像的重建和分割,同时包含一个用于分类的多层感知器(Multi-Layer Perceptron,MLP)。该文利用1 044例患者的数据集(包括449例COVID-19患者、100例正常患者、98例肺癌患者和397例不同病理类型的患者)对所提出的模型进行评估,并对比其他图像分割和分类技术。该模型准确率为0.86,敏感性为0.94,特异性为0.79。
文献[61]采用多任务学习模型,提出一种卷积神经网络模型实现COVID-19检测和患者严重程度的细化。该文训练ResNet50作为分类网络,该网络以轴向切片作为输入,独立提取切片的特征向量,预测肺实质受累的概率,实现对COVID-19的有效检测,而患者严重程度量化任务的实现由两部分组成:左/右肺的分割和病变的分割。肺部的整体分割由一个标准的完全卷积神经网络完成,使用K-means算法对肺部内体素进行聚类,以欧几里得距离作为体素间的度量,完成左/右肺的分割。病变区域的分割则使用基于U-Net结构的网络完成。最后对两个体积的比率进行分级,依次对应不同的严重程度。该模型在COVID-19识别任务上AUC为0.951,在严重程度定量任务上,斯皮尔曼相关性系数达到0.98。
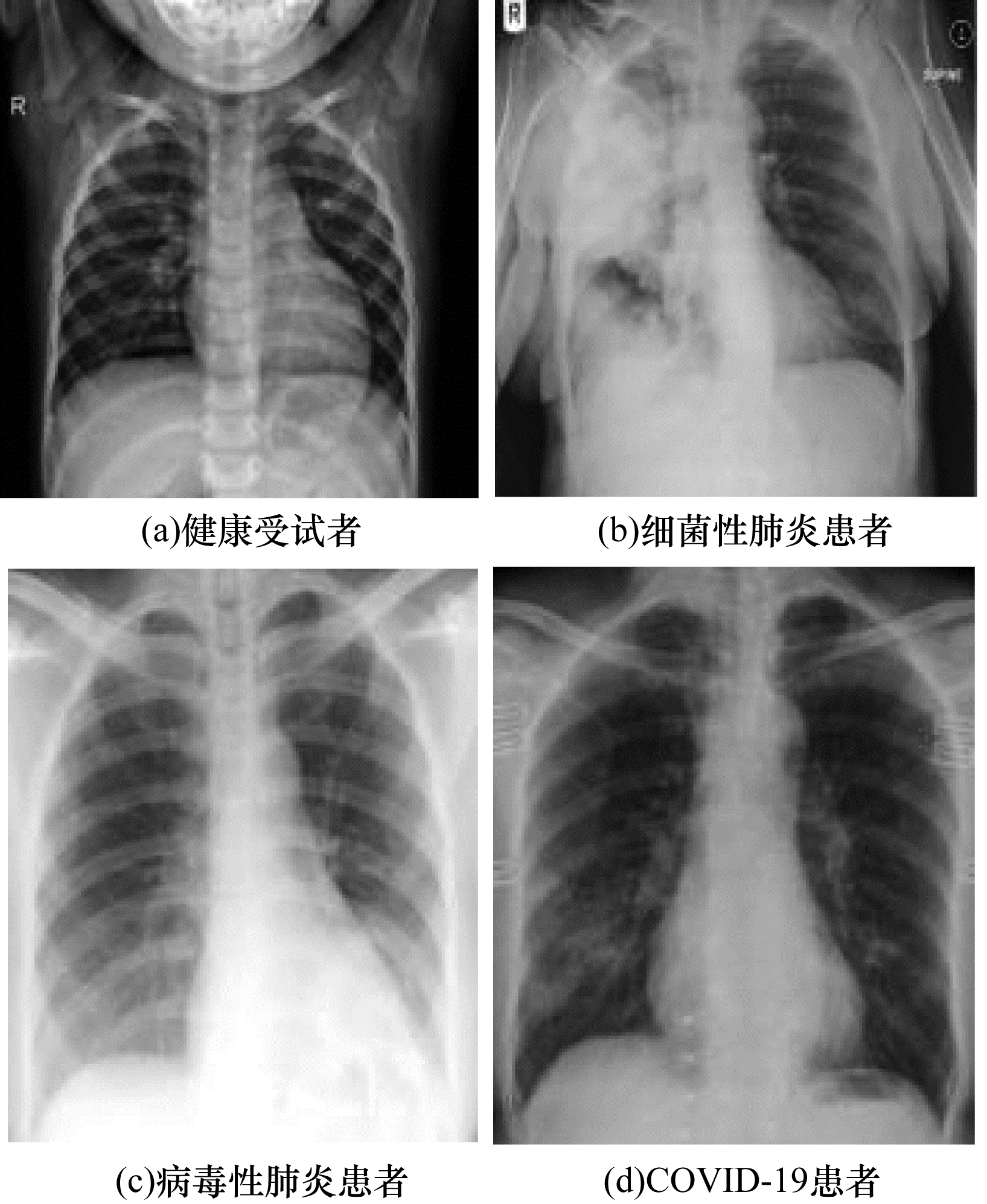
3.2 基于影像数据类型的分类鉴于普通肺炎特别是病毒性肺炎与COVID-19有相似的影像学表现,其鉴别诊断将更有助于临床筛选过程。因此,基于深度学习的COVID-19诊断任务可以概括为健康受试者、其他类型肺炎(细菌性肺炎、病毒性肺炎、社区获得性肺炎等)患者和COVID-19患者的分类任务。不同类型患者在不同影像数据中的典型表现如图 3和图 4所示。
|
Download:
|
| 图 3 胸部X-ray样本图像 Fig. 3 Sample chest X-ray images | |
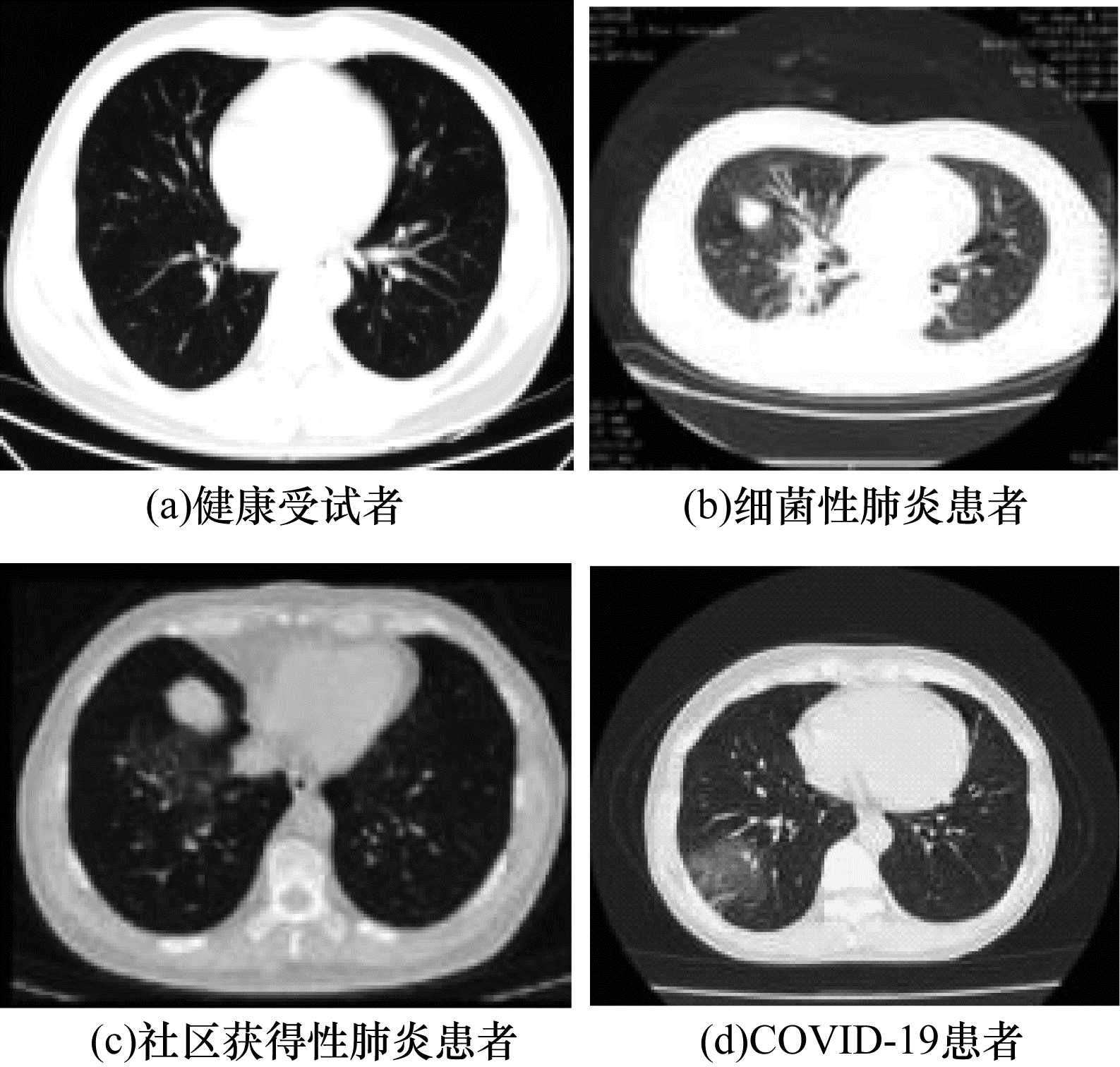
|
Download:
|
| 图 4 胸部CT样本图像 Fig. 4 Sample chest CT images | |
文献[62]提出一种贝叶斯CNN评估COVID-19预测中的诊断不确定性。实验结果表明,在不同的MCDW(Monte-Carlo Drop Weight)下,贝叶斯推理能提高ResNet50V2模型[52]的检测准确率,并且模型的不确定性与预测准确率之间具有强相关性。此外,该文进一步生成显著性图来说明深度网络所关注的位置,从而加深对深度学习结果的理解,做出更明智的决策。
文献[63]提出基于3种不同的深度学习模型,即基于ResNet50、Inception v3和Inception-ResNet v2[64]的COVID-19诊断模型,用于从X-ray图像中检测出COVID-19。通过ROC分析和5-fold交叉验证可以看出:在准确率方面,相对Inception v3的97.0%和Inception-ResNet v2的87%,ResNet50模型获得了98.0%的最高分类性能。
文献[65]提出一个基于EfficientNet[66]的检测诊断模型,使用EfficientNet-B0网络在ImageNet上进行预训练用以提取特征,并且附带异常检测模块和置信分数预测模块。该模型包含两方面的任务:对COVID-19和非COVID-19患者进行分类,并进行异常检测。其中,异常检测任务给出异常分数,以优化用于分类的COVID-19分数。该模型的敏感性为71.7%,特异性为73.8%,AUC为0.836。
文献[67]提出一种基于ResNet的CNN网络模型(COVID-Net)。该模型对正常、细菌性感染、非COVID-19病毒性感染和COVID-19病毒性感染进行预测,并且满足准确率高于80%和计算复杂度小于2.5亿次乘加运算等设计要求。COVID-Net首先在ImageNet数据集[68]上预训练,然后使用Adam优化器在COVIDx数据集上训练。COVIDx数据集包含45例COVID-19患者、1 203例正常受试者、931例病毒性肺炎患者和660病毒性肺炎患者的X-ray数据。该模型的准确率为83.5%,敏感性为100%,精准率为80%。
文献[69]提出利用迁移学习自动检测COVID-19。首先对近年来用于医学图像分类中最先进(SOTA)的CNN模型进行性能评估,其中包括VGG19[70]、MobleNet v2[71]、Xception[72]、Inception和Inception-ResNet v2。然后通过迁移学习[73]在小型医学图像数据集中检测异常,从而实现对COVID-19的诊断。通过对比不同的CNN结果发现,VGG19和MobileNet v2表现最佳。此外,通过计算分析最佳模型对应混淆矩阵可以看出,VGG19具有更高的准确率,其准确率为97.83%,而MobileNet v2在敏感性方面(99.1%)优于VGG19,因此其被证明是特定分类任务和特定数据样本的有效模型。
以上研究涉及的数据集大多来自两个数据集:COVID-chestxray-dataset[31],包括70张COVID-19患者肺部X-ray图像;KAG数据集[33],包括健康受试者、病毒性肺炎患者和细菌性患者的5 863张X-ray图像。由于COVID-19图像数量有限,不足以评估以上方法的鲁棒性,导致其临床中心应用的可推广性也难以被认可。此外,X-ray作为最常用的医学成像方式之一,被广泛用于为放射科医生提供证据,但通常被认为不如胸部CT图像敏感。值得注意的是,最近一项研究报告表明[74],COVID-19患者在早期或者轻度症状阶段,X-ray显示正常。因此,研究人员尝试利用CT数据进行COVID-19的诊断。
3.2.2 基于CT的COVID-19诊断模型文献[75]提出了一种弱监督检测诊断模型,利用三维CT图像检测COVID-19。首先,对于每个患者使用预训练模型U-Net分割肺部区域。然后,将分割的3D肺部区域作为三维CNN的输入,以预测感染COVID-19的概率。三维CNN包括以下3个部分:第一部分为主干网络,由1个三维卷积核、1个BN(Batch Normalization)层和1个池化层组成;第二部分由2个残差块构成;第三部分为渐进式分类器,包含3个卷积层和1个全连接层。在该工作中,收集的542名受试者(313例COVID-19、229例非COVID-19)的胸部CT图像被用作训练和测试数据。该模型的敏感性为90.7%,特异性为91.1%,AUC为0.959。
文献[76]提出一个用于COVID-19分类和预后分析的全自动深度学习模型。该模型使用DenseNet121[77]作为骨干网络,首先将DenseNet121与FPN相结合完成肺部区域的自动分割,然后利用类DenseNet结构(COVID-19Net)建立分类网络,对分割得到的区域进行分类。该文共收集了7个省市的5 372例CT图像,包括COVID-19数据集和表皮生长因子受体(Epidermal Growth Factor Receptor,EGFR)数据集。其中,4 106例具有EGFR基因突变状态的患者样本用来对模型进行预训练[78],使其学习肺部特征,924例COVID-19患者和342例其他肺炎患者样本则用来进行模型训练和验证。在4个外部验证集中,该模型从其他肺炎(AUC=0.87和0.88)和病毒性肺炎(AUC=0.86)中识别COVID-19取得了良好的效果。
文献[79]采用胸部CT扫描数据集,包含2 685例样本(1 658例COVID-19、1 027例普通肺炎)。该模型首先对所有图像进行预处理,采用VBNet[80]分割感染区域、左/右肺、5个肺叶和18个肺段。然后提取感染大小、位置特征和放射特征等手工特征,并使用最小绝对收缩和选择算子[81]进行特征选择。在此基础上,提出一种感染区域大小感知的随机森林方法,将受试者自动分为不同感染病灶大小的分组,并在每组中进行随机森林分类。实验结果表明,该方法对于感染大小在0.01%~10%中等范围内的病例取得了较大的性能裕度,而对小范围感染的患者被识别的敏感性较低。在5-fold交叉验证下,该模型敏感性为90.7%,特异性为83.3%,准确率为87.9%。
文献[82]通过神经结构搜索(NAS)设计一个轻量级的三维模型,结合CT图像检测进行COVID-19诊断。该模型首先建立搜索空间,定义神经结构的设计原则。然后选择搜索策略,相关研究表明,随机搜索比其他许多方法更具竞争力[83-85]。最后选择准确率方面排名靠前的模型进行对比。作者将通过NAS搜索得到的最佳模型命名为MNas3DNet。实验结果表明,相对基线3D模型,MNas3DNet模型尺寸更小。此外,其中MNas3DNet41能实现SOTA级别的性能表现,相应的准确率为87.14%,F1评分为87.25%,AUC为0.957。
文献[86]提出一种基于混合密度网络模型的深度双向长短时记忆网络(DBM),并结合自适应差分进化(MADE)算法[87]构建MADE-DBM模型。该模型可以有效避免手工调整超参数的繁琐过程,适用于COVID-19实时分类系统。该文采用SARS-CoV-2 CT-scan数据集,将训练集和测试集按3∶2的比例进行划分,并对提出的模型与其他SOTA模型分别进行二分类和三分类的性能比较。实验结果表明,该模型在AUC、敏感性、特异性和准确率等指标上相较于其他模型均获得了最佳表现。对于二分类,其AUC达到0.983,敏感性达到0.989,准确率达到98.4%。
文献[39]介绍公开数据集COVID-CT-set,并且提出一种快速、准确的全自动系统用于从CT图像中检测出COVID-19患者的图像。该系统主要流程如下:在第一阶段运行优化的图像处理算法,以丢弃肺部内部无法正常显示的CT图像。在下一阶段提出一种基于ResNet50V2改进的深度卷积网络,并通过特征金字塔网络进行增强,对所选CT图像进行分类。最后利用特征可视化算法对分类后的图像进行处理,以显示图像中的感染区域。实验结果表明,该系统能够以专业医生5倍的识别速度,达到98.49%的准确率。
4 模型性能对比及分析上文对数据集、评价指标和基于深度学习技术的COVID-19检测诊断模型进行了详细介绍。下文将从骨干网络、数据集、数据类型、性能表现、分类种类和开源情况6个维度,分析比较现有基于深度学习的COVID-19检测诊断模型,具体见表 2。

|
下载CSV 表 2 不同COIVID-19诊断模型比较 Table 2 Comparison of different COVID-19 diagnostic models |
从表 2可以得出以下结论:
1)对骨干网络进行分析可知:COVID-19检测诊断模型的网络结构包含分割网络(U-Net、UNet++和VBNet等)和分类网络(ResNet系列、DenseNet系列、Inception系列等),由于COVID-19患者在不同阶段表现出不同的影像学特征,因此可以使用分割网络对ROI区域进行分割提取;同时可以借助分类网络学习COVID-19患者特有的影像学特征,从而有效区分COVID-19、其他肺炎和正常受试者;此外,研究者借助辅助手段(如FPN、注意力机制、决策树、Lasso等)可有效提高模型的性能。
2)对数据集进行分析可知:多数研究工作的COVID-19数据集样本数量范围从几十到几百不等,大部分研究者采用迁移学习,并基于小样本COVID-19数据集进行检测诊断(如文献[67, 75, 79]);此外,目前只有极少量公开可用的大型COVID-19影像数据集(如CC-CCII数据集、MosMedData数据集、SARS-CoV-2 CT-scan数据集、COVID-CT-set数据集),大部分研究人员采用私有数据集(如文献[45, 49, 54, 58]),由此也导致部分模型的泛化能力较弱,对特定数据集有一定的偏移性,因此,无法在相对公平的条件下通过比较不同数据集下的实验结果来评价模型性能优劣。
3)对影像类型进行分析可知:科研工作者倾向基于CT图像的COVID-19诊断(如文献[42, 45, 50]),一方面,因为X-ray相对CT图像敏感性更低;另一方面,CT图像数据除了二维类型外,还包含更多深层次三维特征信息,CNN在图像处理方面的出色表现,本质上源于其强大的特征提取能力,因此,CNN能够从三维CT图像数据中获取更多的特征信息,从而提高诊断的效果。
4)对模型性能进行分析可知:文献[63]提出的ResNet50模型取得了最高的SPE=1.000;文献[67, 51]提出的ResNet模型和UNet++取得了最高的SEN=1.000;文献[56]提出的UNet和ResNet50多任务模型取得了最高的AUC=0.996;文献[39]提出的ResNet50 v2模型取得了最高的ACC=0.985。需要注意的是,该对比缺少一定的指导价值,存在诸多不一致因素,比如不同的验证方式(包括回顾性(内部)验证和前瞻性(外部)验证)、不同的分类级别(包括图像级别和病例级别)、不同的置信区间(Confidence Interval,CI)(大多取CI=95%)、不同的AUC类型(包括PR AUC和ROC AUC)、不同的数据集以及不同的分类种类等。
5)对分类种类分析可知:上述模型的COVID-19分类任务可归纳为二分类(COVID-19与non COVID-19)、三分类(COVID-19、其他肺炎和正常)以及四分类(COVID-19、其他病毒性肺炎、细菌性肺炎和正常)。
6)对开源情况进行分析可知:只有少量模型开源(如文献[39, 49, 59, 75])。由于各个模型之间进行直接比较缺乏一定的公正性,后续可以通过统一的数据集建立相对公平的环境,对目前现有的开源模型进行比较实验(如文献[82]),因此对数据集的统一完善以及开源模型的改进显得至关重要。
综上,现有基于深度学习的COVID-19诊断模型从不同角度进行新冠肺炎的检测诊断,并取得了不错的表现。这些模型不仅能够完成对ROI区域的自动分割和自动标注,有效减少医务人员的人工操作,而且还能对COVID-19患者进行快速筛选,辅助专业人士进行高效诊断。
5 应用实例介绍目前,多种基于深度学习的COVID-19检测诊断模型相继被提出,并经过不断的优化完善,性能已可满足实际应用场景下的临床应用需求。特别是利用CT图像辅助诊断COVID-19的肺炎AI影像辅助产品,极大提高了临床诊断效率,在COVID-19的防控工作中发挥了举足轻重的作用。下文将介绍在抗疫场景下表现出色的人工智能应用系统。
阿里达摩院医疗AI团队基于最新的诊疗方案,包括钟南山院士等多个权威团队发表的关于新冠肺炎患者临床特征的工作[88-90],与浙大一附院、万里云、长远佳和古珀医院等多家机构开展合作,率先突破了训练数据不足的局限,其基于5 000多个病例的CT影像样本数据学习、训练样本的病灶纹理,研发了全新的AI算法模型。该技术能够在20 s内对患者的肺部CT图像作出分析和判读,从而为临床医生的诊断提供有力依据,准确率达到96%以上。此外,该技术还可计算病灶部位的比例,量化、预测病症的轻重程度,大幅度提升诊断效率,为患者的治疗争取宝贵时间。特别是对未接诊过新冠肺炎病例或低年资医生,可提供有效的诊断鉴别提示信息。同时,阿里达摩院还与阿里云共同研发了辅助诊断算法,该算法可以根据患者的基本信息、症状、实验室检查结果、流行病学史、影像报告等多维信息,进一步辅助医生制定科学的治疗方案。
依图胸部CT的COVID-19智能评价系统[91]由上海市公共卫生临床中心和依图医疗合作开发,其作为业界首个COVID-19智能影像评价系统于2020年1月28日上线。该系统可实现前中后期的全流程辅助诊断,并采用创新的AI全肺定量分析技术,提升诊断的速度和精度,帮助医生进行快速检出、定量检测和疗效评价。该系统具体功能和性能为:
1)智能检出:肺炎病变检出率敏感性达97.3%,特异性达99%。
2)智能分析:对肺炎严重程度进行量化评价,在2 s~3 s内完成定量分析。
3)智能随访:智能随访分析患者病程,精准匹配历史影像,自动分析病情的转移和发展。
钟南山等专家通过召集支援武汉一线工作富有临床经验的医师,并联合统计学、生物医学工程和信息技术领域的专家,开发了基于物联网医学技术的COVID-19智能诊治辅助程序,简称nCapp[92]。该程序主要包括以下3个方面的功能:
1)智能辅助诊断:根据提供的数据、问卷回复以及检查结果自动进行确诊,并对疑似和可疑的病例进行归类;根据患者病情的严重程度,区分为轻型、普通型、重型和危重型;建立实时更新的COVID-19样例数据库,通过最新病例数据对智能诊断的算法模型进行实时优化升级,提升诊断的准确率。
2)智能指导治疗:对不同类型患者制定不同的治疗方案,追踪COVID-19愈后患者情况,进行长期随访管理。
3)智能数据监测:自主监控上传至云服务器数据的可靠性与真实性,保证nCapp能够有效地辅助专家进行诊断。
张康教授团队研发的COVID-19智慧筛查、诊断与预测系统,可以对大量疑似肺炎病人进行快速筛查、辅助诊断和住院临床分级预警,实现对COVID-19病人的全生命周期管理。该团队基于五十万份临床影像学大数据[93-94],运用深度学习[95]、迁移学习[96]和语义分割[97-98]等多种AI前沿技术,开发基于胸部CT和X-ray的新冠肺炎AI辅助诊断系统[26]。该系统可在20 s内完成检测及诊断过程,且诊断准确率达90%以上。此外,其还具有病情严重程度分级和重症危重症预测功能,可对胸部CT图像每一层面的小结节、磨玻璃影和实变进行自动识别、标注及定量分析,通过患者的吸氧频率、血氧饱和度、酸碱平衡、肝功能、凝血功能等信息,综合预测病人发展为重症、危重症的概率和时间,有利医生进行及时干预,降低患者死亡率。
6 未来研究方向现有多数基于深度学习的COVID-19检测诊断模型都是基于小样本数据集训练得到的,这可能会导致结果的过拟合。因此,为提高模型性能,使其能够更好地辅助临床诊断,COVID-19数据集的质量和数量都有待进一步提高。在数量方面,需要联合多家医院和研究机构,实现资源共享以扩大样本量;在质量方面,要求所有的影像数据都是规范的和标准的,并需要对这些影像数据进行精确标记。值得注意的是,影像数据中不完整、不精确的标记,将显著降低诊断模型性能,而基于弱监督深度学习技术的COVID-19诊断模型可以有效规避噪声数据(或低质量数据)的影响,这将是未来诊断模型一个较好的发展方向。此外,数据的人工标注往往费时费力,研究者可以尝试利用无监督和自监督[99-100]等方法构建COVID-19诊断模型。另一方面,新冠肺炎诊断不应仅局限于COVID-19和非COVID-19之间的分类,而应扩展至细粒度或者精细化研究[101-102],由此分析得出患者的感染患病等级。与此同时,当下一些热门的方法同样值得借鉴,如多模态数据分析[103-104]、知识图谱[105]和自动机器学习[106-107]等。
7 结束语本文结合基于深度学习的COVID-19检测诊断模型研究现状,在影像数据类型和模型任务方面介绍目前性能较为突出的分类/分割方法并进行整理和评估,对模型性能集中在6个不同维度进行分析。随着相关检测诊断模型研究不断发展,效果显著的模型被陆续提出并被成功应用到抗击COVID-19的第一线,这对抑制COVID-19大规模流行具有重大意义。同时,针对该研究领域构建的影像数据集、常用评价指标和代表性检测诊断模型,对于未来抗击其他传染性疾病同样具有重要的参考价值。后续将对基于多模态知识计算的新冠肺炎病灶区域分割算法进行深入研究,促进多模态融合和知识图谱等技术在医学领域的应用与发展。
| [1] |
WHO. Naming the coronavirus disease(COVID-19) and the virus that causes it[EB/OL]. [2021-01-25]. https://www.who.int/emerg-encies/diseases/novel-coronavirus-2019/technical-guidance.
|
| [2] |
FU Bingjie, ZHANG Hao, YOU Xingpan, et al. Early COVID-19 and common pneumonia: clinical and imaging differential diagnosis[J]. Journal of Chongqing Medical University, 2020, 45(7): 998-1003. (in Chinese) 付彬洁, 张浩, 游兴攀, 等. 早期新型冠状病毒肺炎与普通型肺炎的临床及影像学鉴别诊断[J]. 重庆医科大学学报, 2020, 45(7): 998-1003. |
| [3] |
Johns Hopkins University. COVID-19 dashboard by the center for systems science and engineering at Johns Hopkins university[EB/OL]. [2021-01-25]. https://coronavirus.jhu.edu/map.html.
|
| [4] |
CHAN J F, YUAN S F, KOK K, et al. A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster[J]. The Lancet, 2020, 395(10223): 514-523. DOI:10.1016/S0140-6736(20)30154-9 |
| [5] |
ZHU Na, ZHANG Dingyu, WANG Wenling, et al. A novel coronavirus from patients with pneumonia in China, 2019[J]. New England Journal of Medicine, 2020, 382(8): 727-733. DOI:10.1056/NEJMoa2001017 |
| [6] |
CHEN Nanshan, ZHOU Min, DONG Xuan, et al. Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study[J]. The Lancet, 2020, 395(10223): 507-513. DOI:10.1016/S0140-6736(20)30211-7 |
| [7] |
YANG Xiaobo, YU Yuan, XU Jiqian, et al. Clinical course and outcomes of critically ill patients with SARS-CoV-2 pneumonia in Wuhan, China: a single-centred, retrospective, observational study[J]. The Lancet Respiratory Medicine, 2020, 8(5): 475-481. DOI:10.1016/S2213-2600(20)30079-5 |
| [8] |
WHO. Q&A on coronaviruses(COVID-19)[EB/OL]. [2021-01-25]. https://www.who.int/emergencies/diseases/novel-coronavirus-2019/question-and-answers-hub/q-a-detail/q-a-coronaviruses.
|
| [9] |
CHIN A, CHU J L, PERERA M, et al. Stability of SARS-CoV-2 in different environmental conditions[J]. The Lancet Microbe, 2020, 1(1): 1-5. DOI:10.1016/S2666-5247(20)30016-1 |
| [10] |
SILVERMAN J D, HUPERT N, WASHBURNE A D. Using influenza surveillance networks to estimate state-specific prevalence of SARS-CoV-2 in the United States[J]. Science Translational Medicine, 2020, 12(554): 1-11. |
| [11] |
WU Aiping, PENG Yousong, HUANG Baoying, et al. Genome composition and divergence of the novel coronavirus(2019-nCoV) originating in China[J]. Cell Host & Microbe, 2020, 27(3): 325-328. |
| [12] |
ZHANG Wei, HOU Wei, LI Tongzeng, et al. Clinical characteristics analysis of 74 cases of COVID-19[J]. Journal of Capital Medical University, 2020, 41(2): 161-167. (in Chinese) 张维, 侯维, 李侗曾, 等. 74例新型冠状病毒肺炎临床特征分析[J]. 首都医科大学学报, 2020, 41(2): 161-167. DOI:10.3969/j.issn.1006-7795.2020.02.003 |
| [13] |
GUAN Weijie, NI Zhengyi, HU Yu, et al. Clinical characteristics of coronavirus disease 2019 in China[J]. New England Journal of Medicine, 2020, 382(18): 1708-1720. DOI:10.1056/NEJMoa2002032 |
| [14] |
WU Z, McGOOGAN J M. Characteristics of and important lessons from the Coronavirus Disease 2019(COVID-19) outbreak in China: summary of a report of 72314 cases from the Chinese center for disease control and prevention[J]. JAMA, 2020, 323(13): 1239-1242. DOI:10.1001/jama.2020.2648 |
| [15] |
HOPE M D, RAPTIS C A, HENRY T S. Chest computed tomography for detection of Coronavirus Disease 2019(COVID-19): don't rush the science[J]. Annals of Internal Medicine, 2020, 173(2): 1-3. |
| [16] |
LAUER S A, GRANTZ K H, BI Q F, et al. The incubation period of Coronavirus Disease 2019(COVID-19) from publicly reported confirmed cases: estimation and application[J]. Annals of Internal Medicine, 2020, 172(9): 577-582. DOI:10.7326/M20-0504 |
| [17] |
WHO. Clinical management of severe acute respiratory infection when novel coronavirus(2019-nCoV) infection is suspected: interim guidance[EB/OL]. [2021-01-25]. https://apps.who.int/ir-is/handle/10665/330893.
|
| [18] |
LI Shixue, SHAN Ying. Review of research progress in COVID-19[J]. Journal of Shandong University(Medical Edition), 2020, 58(3): 19-25. (in Chinese) 李士雪, 单莹. 新型冠状病毒肺炎研究进展述评[J]. 山东大学学报(医学版), 2020, 58(3): 19-25. |
| [19] |
FU Hang, XU Huanyan, ZHANG Na, et al. Association between clinical, laboratory and CT characteristics and RT-PCR results in the follow-up of COVID-19 patients[EB/OL]. [2021-01-25]. https://www.med-rxiv.org/content/10.1101/2020.03.19.20038315v1.full.pdf.
|
| [20] |
XIE Xingzhi, ZHONG Zheng, ZHAO Wei, et al. Chest CT for typical 2019-nCoV pneumonia: relationship to negative RT-PCR testing[J]. Radiology, 2020, 296(2): 1-5. |
| [21] |
National Health Commission of the People's Republic of China. Diagnosis and treatment protocol for COVID-19(trial version7)[EB/OL]. [2021-01-25]. http://en.nhc.gov.cn/2020-03/29/c_78469.htm.
|
| [22] |
AI Tao, YANG Zhenlu, HOU Hongyan, et al. Correlation of chest CT and RT-PCR testing in Coronavirus Disease 2019(COVID-19) in China: a report of 1014 cases[J]. Radiology, 2020, 296(2): 1-9. |
| [23] |
SHI Heshui, HAN Xiaoyu, JIANG Nanchuan, et al. Radiological findings from 81 patients with COVID-19 pneumonia in Wuhan, China: a descriptive study[J]. The Lancet Infectious Diseases, 2020, 20(4): 425-434. DOI:10.1016/S1473-3099(20)30086-4 |
| [24] |
CHUNG M, BERNHEIM A, MEI X Y, et al. CT imaging features of 2019 novel Coronavirus(2019-nCoV)[J]. Radiology, 2020, 295(1): 202-207. DOI:10.1148/radiol.2020200230 |
| [25] |
ZHONG Feiyang, ZHANG Hanfei, WANG Binchen, et al. CT imaging findings in novel coronavirus pneumonia[J]. Medical Journal of Wuhan University, 2020, 41(3): 345-348. (in Chinese) 钟飞扬, 张寒菲, 王彬宸, 等. 新型冠状病毒肺炎的CT影像学表现[J]. 武汉大学学报(医学版), 2020, 41(3): 345-348. |
| [26] |
YE Zheng, ZHANG Yun, WANG Yi, et al. Chest CT manifestations of new Coronavirus Disease 2019(COVID-19): a pictorial review[J]. European Radiology, 2020, 30: 4381-4389. DOI:10.1007/s00330-020-06801-0 |
| [27] |
RODRIGUEZ-MORALES A J, CARDONA-OSPINA J A, GUTIÉRREZ-OCAMPO E, et al. Clinical, laboratory and imaging features of COVID-19:a systematic review and meta-analysis[J]. Travel Medicine and Infectious Disease, 2020, 34: 1-19. DOI:10.1016/j.tmaid.2020.101623 |
| [28] |
ZHANG Nan, ZOU Mingyu, ZHOU Shu. Application of CT low-dose scan combined with AI-assisted diagnosis system in the examination of COVID-19[J]. Chinese Medical Equipment Journal, 2020, 41(5): 9-11. (in Chinese) 张楠, 邹明宇, 周姝. CT低剂量扫描结合AI辅助诊断系统在新型冠状病毒肺炎检查中的应用[J]. 医疗卫生装备, 2020, 41(5): 9-11. |
| [29] |
RAJPURKAR P, IRVIN J, ZHU K, et al. CheXNet: radiologist-level pneumonia detection on chest X-rays with deep learning[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/1711.05225.pdf.
|
| [30] |
ZECH J R, BADGELEY M A, LIU M, et al. Variable generalization performance of a deep learning model to detect pneumonia in chest radiographs: a cross-sectional study[J]. PLoS Medicine, 2018, 15(11): 1-17. |
| [31] |
ZHANG Kang, LIU Xiaohong, SHEN Jun, et al. Clinically applicable AI system for accurate diagnosis, quantitative measurements and prognosis of COVID-19 pneumonia using computed tomography[J]. Cell, 2020, 181(6): 1423-1433. DOI:10.1016/j.cell.2020.04.045 |
| [32] |
COHEN J P, MORRISON P, DAO L, et al. COVID-19 image data collection: prospective predictions are the future[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2006.11988.pdf.
|
| [33] |
Kaggle. Chest X-Ray images (pneumonia)[EB/OL]. [2021-01-25]. https://www.kaggle.com/paultimothymooney/chest-xray-pneumonia.
|
| [34] |
YANG Xingyi, HE Xuehai, ZHAO Jinyu, et al. COVID-CT-Dataset: a CT scan dataset about COVID-19[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2003.13865.pdf.
|
| [35] |
MA Jun, GE Cheng, WANG Yixin, et al. COVID-19 CT lung and infection segmentation dataset[EB/OL]. [2021-01-25]. https://doi.org/10.5281/zenodo.3757476.
|
| [36] |
MOROZOV S P, ANDREYCHENKO A E, PAVLOV N A, et al. MosMedData: chest CT scans with COVID-19 related findings dataset[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2005.06465.pdf.
|
| [37] |
SOARES E, ANGELOV P, BIASO S, et al. SARS-CoV-2 CT-scan dataset: a large dataset of real patients CT scans for SARS-CoV-2 identification[EB/OL]. [2021-01-25]. https://www.medrxiv.org/content/10.1101/2020.04.24.20078584v3.full.pdf.
|
| [38] |
RAHIMZADEH M, ATTAR A, SAKHAEI S M. A fully automated deep learning-based network for detecting COVID-19 from a new and large lung CT scan dataset[EB/OL]. [2021-01-25]. https://www.med-rxiv.org/content/10.1101/2020.06.08.20121541v2.full.pdf.
|
| [39] |
PENG Y F, TANG Y X, LEE S, et al. COVID-19-CT-CXR: a freely accessible and weakly labeled chest X-ray and CT image collection on COVID-19 from biomedical literature[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2006.06177.pdf.
|
| [40] |
GOUTTE C, GAUSSIER E. A probabilistic interpretation of precision, recall and F-score, with implication for evaluation[C]//Proceedings of European Conference on Information Retrieval. Berlin, Germany: Springer, 2005: 345-359.
|
| [41] |
SOKOLOVA M, JAPKOWICZ N, SZPAKOWICZ S. Beyond accuracy, F-score and ROC: a family of discriminant measures for performance evaluation[C]//Proceedings of Australasian Joint Conference on Artificial Intelligence. Washington D.C., USA: IEEE Press, 2006: 1015-1021.
|
| [42] |
WANG Shuai, KANG Bo, MA Jinlu, et al. A deep learning algorithm using CT images to screen for corona virus disease(COVID-19)[EB/OL]. [2021-01-25]. https://www.medrxiv.org/content/medrxiv/early/2020/02/17/2020.02.14.20023028.full.pdf.
|
| [43] |
SZEGEDY C, LIU W, JIA Y Q, et al. Going deeper with convolutions[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2015: 1-9.
|
| [44] |
HASTIE T, ROSSET S, ZHU J, et al. Multi-class AdaBoost[J]. Statistics and Its Interface, 2009, 2(3): 349-360. DOI:10.4310/SII.2009.v2.n3.a8 |
| [45] |
SONG Ying, ZHENG Shuangjia, LI Liang, et al. Deep learning enables accurate diagnosis of novel coronavirus(COVID-19) with CT images[EB/OL]. [2021-01-25]. https://www.medrxiv.org/content/10.1101/2020.02.23.20026930v1.full.pdf.
|
| [46] |
HE Kaiming, ZHANG Xiangyu, REN Shaoqing, et al. Deep residual learning for image recognition[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2016: 770-778.
|
| [47] |
LIN T Y, DOLLAR P, GIRSHICK R, et al. Feature pyramid networks for object detection[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2017: 2117-2125.
|
| [48] |
FU Jianlong, ZHENG Heliang, MEI Tao. Look closer to see better: recurrent attention convolutional neural network for fine-grained image recognition[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2017: 4438-4446.
|
| [49] |
WANG Xin, KONG Bin, SONG Qi, et al. Artificial intelligence distinguishes COVID-19 from community acquired pneumonia on chest CT[J]. Radiology, 2020, 296(2): 1-17. |
| [50] |
CHEN Jun, WU Lian, ZHANG Jun, et al. Deep learning-based model for detecting 2019 novel coronavirus pneumonia on high-resolution computed tomography: a prospective study[EB/OL]. [2021-01-25]. https://www.medrxiv.org/content/10.1101/2020.02.25.20021568v2.full.pdf.
|
| [51] |
ZHOU Z W, SIDDIQUEE M, TAJBAKHSH N, et al. UNet++: a nested U-Net architecture for medical image segmentation[M]//CARDOSO J, ARBEL T, CARNEIRO G, et al. Deep learning in medical image analysis and multimodal learning for clinical decision support. Berlin, Germany: Springer, 2018: 3-11.
|
| [52] |
RONNEBERGER O, FISCHER P, BROX T. U-Net: convolutional networks for biomedical image segmentation[M]//OURSELIN S, JOSKOWICZ L, SABUNCU M R, et al. Medical image computing and computer-assisted intervention. Berlin, Germany: Springer, 2015: 234-241.
|
| [53] |
JAISWAL A, GIANCHANDANI N, SINGH D, et al. Classification of the COVID-19 infected patients using DenseNet201 based deep transfer learning[EB/OL]. (2020-06-03)[2021-01-25]. https://www.tandfonline.com/doi/full/10.1080/07391102.2020.1788642.
|
| [54] |
BUTT C, GILL J, CHUN D, et al. Deep learning system to screen Corona-Virus Disease 2019 pneumonia[EB/OL]. (2020-04-22)[2021-01-25]. http://link.springer.com/content/pdf/10.1007/s10489-020-01714-3.pdf.
|
| [55] |
HARA K, KATAOKA H, SATOH Y. Learning spatio-temporal features with 3D residual networks for action recognition[C]//Proceedings of IEEE International Conference on Computer Vision. Washington D.C., USA: IEEE Press, 2017: 3154-3160.
|
| [56] |
GOZES O, FRID-ADAR M, GREENSPAN H, et al. Rapid AI development cycle for the coronavirus(COVID-19) pandemic: initial results for automated detection & patient monitoring using deep learning CT image analysis[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2003.05037.pdf.
|
| [57] |
HE Kaiming, ZHANG Xiangyu, REN Shaoqing, et al. Identity mappings in deep residual networks[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/1603.05027.pdf.
|
| [58] |
JIN Shuo, WANG Bo, XU Haibo, et al. AI-assisted CT imaging analysis for COVID-19 screening: building and deploying a medical AI system in four weeks[EB/OL]. [2021-01-25]. https://www.medrxiv.org/content/10.1101/2020.03.19.20039354v1.full.pdf.
|
| [59] |
AMYAR A, MODZELEWSKI R, RUAN S. Multi-task deep learning based CT imaging analysis for COVID-19: classification and segmentation[EB/OL]. [2021-01-25]. https://www.medrxiv.org/content/10.1101/2020.04.16.20064709v1.full.pdf.
|
| [60] |
CARUANA R. Multitask learning[J]. Machine Learning, 1997, 28(1): 41-75. DOI:10.1023/A:1007379606734 |
| [61] |
GONCHAROV M, PISOV M, SHEVTSOV A, et al. CT-based COVID-19 triage: deep multitask learning improves joint identification and severity quantification[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2006.01441.pdf.
|
| [62] |
GHOSHAL B, TUCKER A. Estimating uncertainty and interpretability in deep learning for coronavirus(COVID-19) detection[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2003.10769.pdf.
|
| [63] |
NARIN A, KAYA C, PAMUK Z. Automatic detection of coronavirus disease(COVID-19) using X-ray images and deep convolutional neural networks[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2003.10849.pdf.
|
| [64] |
SZEGEDY C, IOFFE S, VANHOUCKE V, et al. Inception-v4, inception-ResNet and the impact of residual connections on learning[C]//Proceedings of the 31st AAAI Conference on Artificial Intelligence. San Francisco, USA: AAAI, 2017: 4278-4284.
|
| [65] |
ZHANG Jianpeng, XIE Yutong, LIAO Zhibin, et al. Viral pneumonia screening on chest X-ray images using confidence-aware anomaly detection[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2003.12338.pdf.
|
| [66] |
TAN M, LE Q V. EfficientNet: rethinking model scaling for convolutional neural networks[C]//Proceedings of International Conference on Machine Learning. Long Beach, USA: IMLS, 2019: 6105-6114.
|
| [67] |
WANG L, WONG A. COVID-Net: a tailored deep convolutional neural network design for detection of COVID-19 cases from chest X-ray images[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2003.09871.pdf.
|
| [68] |
DENG J, DONG W, SOCHER R, et al. ImageNet: a large-scale hierarchical image database[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2009: 248-255.
|
| [69] |
APOSTOLOPOULOS I D, MPESIANA T A. COVID-19:automatic detection from X-ray images utilizing transfer learning with convolutional neural networks[J]. Physical and Engineering Sciences in Medicine, 2020, 43(2): 635-640. DOI:10.1007/s13246-020-00865-4 |
| [70] |
SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/1409.1556.pdf.
|
| [71] |
HOWARD A G, ZHU M L, CHEN B, et al. MobileNets: efficient convolutional neural networks for mobile vision applications[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/1704.04861.pdf.
|
| [72] |
CHOLLET F. Xception: deep learning with depthwise separable convolutions[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2017: 1251-1258.
|
| [73] |
PAN S J, TSANG I W, KWOK J T, et al. Domain adaptation via transfer component analysis[J]. IEEE Transactions on Neural Networks, 2011, 22(2): 199-210. DOI:10.1109/TNN.2010.2091281 |
| [74] |
WONG H Y F, LAM H Y S, FONG A H, et al. Frequency and distribution of chest radiographic findings in COVID-19 positive patients[J]. Radiology, 2020, 296: 72-78. DOI:10.1148/radiol.2020201160 |
| [75] |
ZHENG Chuansheng, DENG Xianbo, FU Qing, et al. Deep learning-based detection for COVID-19 from chest CT using weak label[EB/OL]. [2021-01-25]. https://www.medrxiv.org/content/medrxiv/early/2020/03/26/2020.03.12.20027185.full.pdf.
|
| [76] |
WANG Shuo, ZHA Yunfei, LI Weimin, et al. A fully automatic deep learning system for COVID-19 diagnostic and prognostic analysis[J]. European Respiratory Journal, 2020, 56(1): 1-31. |
| [77] |
HUANG G, LIU Z, MAATEN L V D, et al. Densely connected convolutional networks[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2017: 4700-4708.
|
| [78] |
WANG Shuo, SHI Jingyun, YE Zhaoxiang, et al. Predicting EGFR mutation status in lung adenocarcinoma on computed tomography image using deep learning[J]. European Respiratory Journal, 2019, 53(3): 1-45. |
| [79] |
SHI Feng, XIA Liming, SHAN Fei, et al. Large-scale screening of COVID-19 from community acquired pneumonia using infection size-aware classification[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2003.09860.pdf.
|
| [80] |
SHAN Fei, GAO Yaozong, WANG Jun, et al. Lung infection quantification of COVID-19 in CT images with deep learning[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2003.04655.pdf.
|
| [81] |
PARK T, CASELLA G. The Bayesian lasso[J]. Journal of the American Statistical Association, 2008, 103(482): 681-686. DOI:10.1198/016214508000000337 |
| [82] |
HE Xin, WANG Shihao, SHI Shaohuai, et al. Benchmarking deep learning models and automated model design for COVID-19 detection with chest CT scans[EB/OL]. [2021-01-25]. https://www.med-rxiv.org/content/10.1101/2020.06.08.20125963v2.full.pdf.
|
| [83] |
LI L, TALWALKAR A. Random search and reproducibility for neural architecture search[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/1902.07638.pdf.
|
| [84] |
PHAM H, GUAN M Y, ZOPH B, et al. Efficient neural architecture search via parameter sharing[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/1802.03268.pdf.
|
| [85] |
YU K C, SCIUTO C, JAGGI M, et al. Evaluating the search phase of neural architecture search[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/1902.08142.pdf.
|
| [86] |
PATHAK Y, SHUKLA P K, ARYA K V. Deep bidirectional classification model for COVID-19 disease infected patients[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2020, 1: 1-5. |
| [87] |
LIAO Qing, FAN Qinqin, LI Junjun. Translation control of an immersed tunnel element using a multi-objective differential evolution algorithm[J]. Computers & Industrial Engineering, 2019, 130: 158-165. |
| [88] |
CHEN Huijun, GUO Juanjuan, WANG Chen, et al. Clinical characteristics and intrauterine vertical transmission potential of COVID-19 infection in nine pregnant women: a retrospective review of medical records[J]. The Lancet, 2020, 395(10226): 809-915. DOI:10.1016/S0140-6736(20)30360-3 |
| [89] |
HU Zhiliang, SONG Ci, XU Chuanjun, et al. Clinical characteristics of 24 asymptomatic infections with COVID-19 screened among close contacts in Nanjing, China[J]. Science China Life Sciences, 2020, 63: 706-711. DOI:10.1007/s11427-020-1661-4 |
| [90] |
JIANG Fang, DENG Liehua, ZHANG Liangqing, et al. Review of the clinical characteristics of Coronavirus Disease 2019(COVID-19)[J]. Journal of General Internal Medicine, 2020, 35: 1545-1549. DOI:10.1007/s11606-020-05762-w |
| [91] |
McCALL B. COVID-19 and artificial intelligence: protecting health-care workers and curbing the spread[J]. The Lancet Digital Health, 2020, 2(4): 166-167. DOI:10.1016/S2589-7500(20)30054-6 |
| [92] |
BAI Li, YANG Dawei, WANG Xun, et al. Chinese experts' consensus on the Internet of Things-aided diagnosis and treatment of Coronavirus Disease 2019(COVID-19)[J]. Clinical eHealth, 2020, 3: 7-15. DOI:10.1016/j.ceh.2020.03.001 |
| [93] |
OBERMEYER Z, EMANUEL E J. Predicting the future-big data, machine learning, and clinical medicine[J]. The New England Journal of Medicine, 2016, 375(13): 1216-1219. DOI:10.1056/NEJMp1606181 |
| [94] |
WANG C J, NG C Y, BROOK R H. Response to COVID-19 in Taiwan: big data analytics, new technology, and proactive testing[J]. JAMA, 2020, 323(14): 1341-1342. DOI:10.1001/jama.2020.3151 |
| [95] |
SINGH D, KUMAR V, KAUR M. Classification of COVID-19 patients from chest CT images using multi-objective differential evolution-based convolutional neural networks[J]. European Journal of Clinical Microbiology & Infectious Diseases, 2020, 39(7): 1-11. |
| [96] |
MAGHDID H S, ASAAD A T, GHAFOOR K Z, et al. Diagnosing COVID-19 pneumonia from X-ray and CT images using deep learning and transfer learning algorithms[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/2004.00038.pdf.
|
| [97] |
LONG J, SHELHAMER E, DARRELL T. Fully convolutional networks for semantic segmentation[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2015: 3431-3440.
|
| [98] |
GIRSHICK R, DONAHUE J, DARRELL T, et al. Rich feature hierarchies for accurate object detection and semantic segmentation[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2014: 580-587.
|
| [99] |
DOERSCH C, ZISSERMAN A. Multi-task self-supervised visual learning[C]//Proceedings of IEEE International Conference on Computer Vision. Washington D.C., USA: IEEE Press, 2017: 2051-2060.
|
| [100] |
PATHAK D, AGRAWAL P, EFROS A A, et al. Curiosity-driven exploration by self-supervised prediction[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2017: 16-17.
|
| [101] |
YANG Ran, LI Xiang, LIU Huan, et al. Chest CT severity score: an imaging tool for assessing severe COVID-19[J]. Radiology: Cardiothoracic Imaging, 2020, 2(2): 1-8. |
| [102] |
SHI Weiya, PENG Xueqing, LIU Tiefu, et al. Deep learning-based quantitative computed tomography model in predicting the severity of COVID-19: a retrospective study in 196 patients[EB/OL]. [2021-01-02]. https://papers.ssrn.com/sol3/papers.cfm?abstract_id=3546089.
|
| [103] |
ZHANG Wenlu, LI Rongjian, DENG Houtao, et al. Deep convolutional neural networks for multi-modality isointense infant brain image segmentation[J]. NeuroImage, 2015, 108: 214-224. DOI:10.1016/j.neuroimage.2014.12.061 |
| [104] |
HUANG Y W, SHAO L, FRANGI A F. Simultaneous super-resolution and cross-modality synthesis of 3D medical images using weakly-supervised joint convolutional sparse coding[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2017: 6070-6079.
|
| [105] |
ROTMENSCH M, HALPERN Y, TLIMAT A, et al. Learning a health knowledge graph from electronic medical records[J]. Scientific Reports, 2017, 7(1): 1-11. DOI:10.1038/s41598-016-0028-x |
| [106] |
HE Xin, ZHAO Kaiyong, CHU Xiaowen. AutoML: a survey of the state-of-the-art[EB/OL]. [2021-01-25]. https://arxiv.org/pdf/1908.00709.pdf.
|
| [107] |
FAES L, WAGNER S K, FU D J, et al. Automated deep learning design for medical image classification by health-care professionals with no coding experience: a feasibility study[J]. The Lancet Digital Health, 2019, 1(5): 232-242. DOI:10.1016/S2589-7500(19)30108-6 |
 2021, Vol. 47
2021, Vol. 47

