葡萄在生长过程中受到环境、细菌等影响而产生黑腐病、褐斑病、黄叶病和毛毡病等病害,这些病害多数发生在葡萄树的叶片部位,对葡萄的产量和品质造成不利影响。传统凭借经验和病理知识的人工确诊方式存在周期长且耗费人力的问题,而计算机视觉的发展给农作物病害识别带来了新的研究方向[1-2],比如日趋成熟的机器学习[3-5]与深度学习[6-8]为葡萄叶片病害识别提供了新的解决方案。不同程度的同种葡萄病害在病斑的纹理和颜色等方面具有较强的相似性,导致识别不同程度病害的准确率降低。基于深度学习的卷积神经网络能够避免人为设计提取病害特征的问题,为实现葡萄叶片病害识别的自动化,文献[9]采用多类支持向量机对葡萄叶片病害进行分类,平均识别准确率可达到90%。文献[10]采用UnitedModel对4种葡萄叶片病害进行识别,使得识别准确率达到99.17%。文献[11]采用尺度不变特征转换(Scale Invariant Feature Transform,SIFT)特征对叶片序列日期进行匹配得到生长序列图,并通过检测序列图中病斑面积与数量的变化情况诊断叶片病害。但是该方法对叶片姿态要求较为严格,存在一定的局限性。文献[12]使用MobileNet对6种葡萄病害进行识别,获得了87.50%的平均识别准确率。文献[13]采用OTSU算法对葡萄叶片病害区域进行分割,并利用形态学算法改善病害形态,选择Prewitt算子提取病变区域的完整边缘,使用BP神经网络进行病害识别。文献[14]提出一种葡萄叶片病害自动分割识别系统,平均分割准确率和分类准确率可分别达到90%和92%。文献[15]采用全局阈值法提取葡萄病害部位,通过支持向量机对葡萄叶片图像进行分类,并在1 135张葡萄叶片病害测试图像上获得了93.035%的最佳识别准确率。文献[16]提出一种基于图像分析和BPNN的葡萄叶片病害自动检测方法,使得准确率高达91%。
以上传统的机器学习方法需要人工提取病斑特征,对病害表征相似的病害提取特征能力有限,且不同病害通常需要提取不同的特征,因此此类方法不具有普适性。本文在ResNet[17]网络的基础上,通过引入多卷积组合[18]结构和SENet(Squeeze-and-Excitation Networks)[19]来提高网络特征提取能力,并利用构建的识别模型实现对葡萄叶片病害的准确识别。
1 实验数据本文研究的葡萄叶片病害图像由以下2个数据集组成:AI Challenger中葡萄叶片的数据集和从宁夏志辉源石酒庄葡萄酒庄采集的葡萄叶片毛毡病与健康图像数据集,采集时间为2019年5月,在自然光条件下使用小米6X手机拍摄。8种葡萄叶片病害样本图像如图 1所示。

|
Download:
|
| 图 1 8种葡萄叶片病害样本图像 Fig. 1 Sample images of eight grape leaf diseases | |
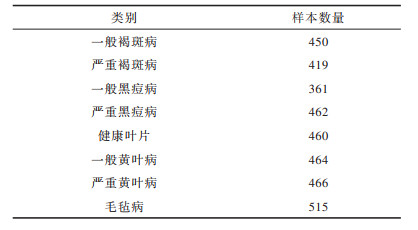
由于AI Challenger中黄叶病一般程度的病害叶片数量较少,为平衡各类别病害样本数量,对黄叶病一般程度的病害叶片进行90°、180°和270°的旋转和明亮度的变化操作,使得数据扩充至原始数据的7倍,扩充后的数据集构成如表 1所示。
|
下载CSV 表 1 实验数据集构成 Table 1 Composition of experimental dataset |
为更好地识别葡萄叶片病害,防止病害图像的非叶片区域影响病害识别而导致识别准确率下降,应先提取葡萄病害图像中的葡萄叶片区域,再对葡萄叶片区域进行病害识别。采用Mask R-CNN[20]对葡萄叶片区域进行提取,并将提取的葡萄叶片输入Multi-Scale ResNet中进行处理和识别。
损失函数能够反映模型与实际数据的差异,且损失函数越小则表明模型效果越好。采用Mask R-CNN提取葡萄叶片时,每一个感兴趣区域(Region of Interest,RoI)的损失函数L如式(1)所示:
| $ L=L\left(\mathrm{c}\mathrm{l}\mathrm{a}\mathrm{s}\mathrm{s}\right)+L\left(\mathrm{b}\mathrm{b}\mathrm{o}\mathrm{x}\right)+L\left(\mathrm{m}\mathrm{a}\mathrm{s}\mathrm{k}\right) $ | (1) |
其中,
| $ L\left(\mathrm{c}\mathrm{l}\mathrm{a}\mathrm{s}\mathrm{s}\right)=-\mathrm{l}\mathrm{n} \; {p}_{u} $ | (2) |
其中,
边框回归的目的是增大输出边框与真实边框之间的IoU(Intersection-over-Union)重叠区域。
| $ L\left(\mathrm{b}\mathrm{b}\mathrm{o}\mathrm{x}\right)=\sum\limits_{i=1}^{4}\mathrm{s}\mathrm{m}\mathrm{o}\mathrm{o}\mathrm{t}{\mathrm{h}}_{{L}_{1}}({t}_{i}^{u}-{v}_{i}) $ | (3) |
其中,
| $ \mathrm{s}\mathrm{m}\mathrm{o}\mathrm{o}\mathrm{t}{\mathrm{h}}_{{L}_{1}}\left(x\right)=\left\{\begin{array}{l}0.5{x}^{2}, \left|x\right|<1\\ \left|x\right|-0.5, \mathrm{其}\mathrm{他}\end{array}\right. $ | (4) |
| $ L\left(\mathrm{m}\mathrm{a}\mathrm{s}\mathrm{k}\right)=\mathrm{S}\mathrm{i}\mathrm{g}\mathrm{m}\mathrm{o}\mathrm{d}(\mathrm{c}\mathrm{l}\mathrm{a}\mathrm{s}\mathrm{s}\_k) $ | (5) |
其中,k表示叶子种类数目。
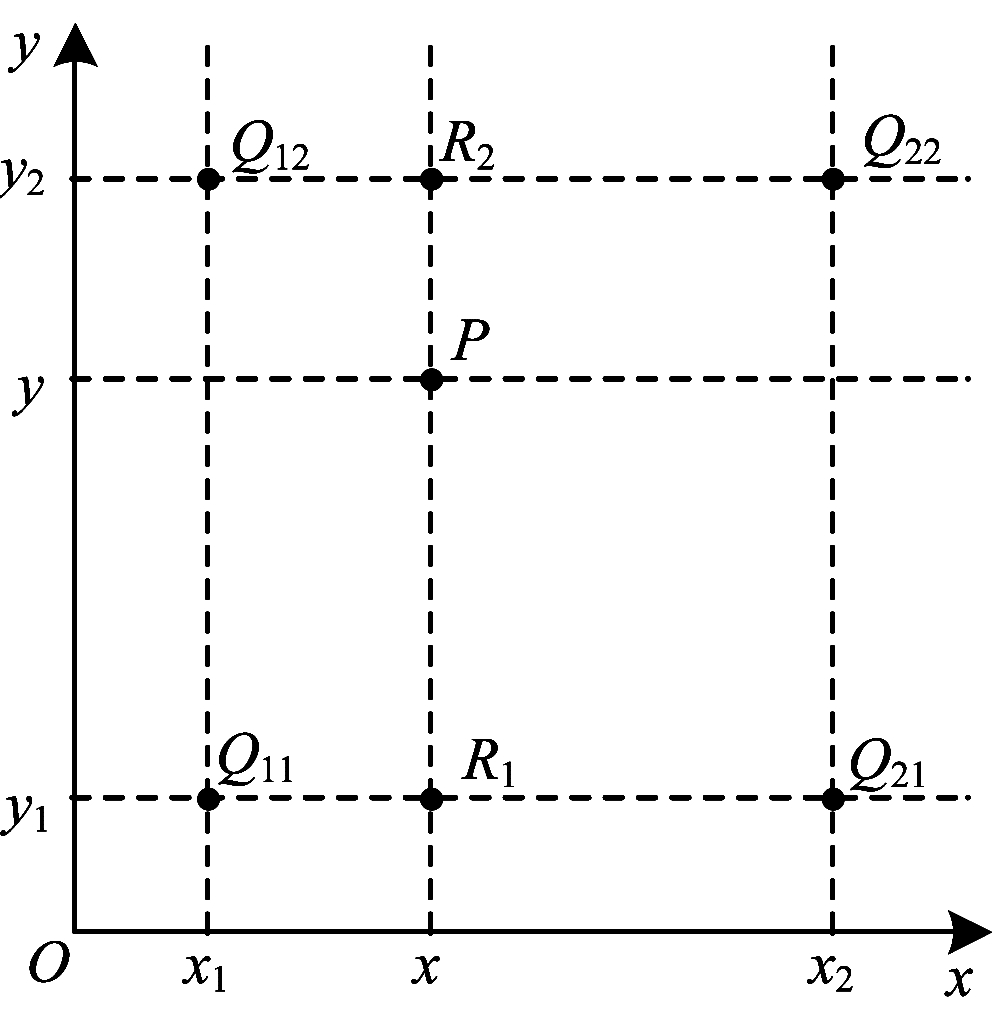
2.2 葡萄叶片提取为更好地提取葡萄叶片,本文利用Mask R-CNN提取葡萄叶片。在提取葡萄叶片之前,由于Mask R-CNN需要统一图像大小作为网络输入,因此选取双线性插值法对输入图像进行处理。双线性内插法示意图如图 2所示,即P点值先在x方向上进行一次线性插值,再在y方向上进行一次线性插值。
|
Download:
|
| 图 2 双线性内插法示意图 Fig. 2 Schematic diagram of bilinear interpolation method | |
综合x和y这2个方向上的双线性插值如式(6)所示:
| $ \begin{array}{l}f(x, y)\approx \frac{f\left({Q}_{11}\right)}{({x}_{2}-{x}_{1})({y}_{2}-{y}_{1})}({x}_{2}-x)({y}_{2}-y)+\\ \;\;\;\;\;\;\; \;\;\;\;\;\;\;\frac{f\left({Q}_{21}\right)}{({x}_{2}-{x}_{1})({y}_{2}-{y}_{1})}(x-{x}_{1})({y}_{2}-y)+\\ \;\;\;\;\;\;\;\;\;\;\;\;\;\; \frac{f\left({Q}_{12}\right)}{({x}_{2}-{x}_{1})({y}_{2}-{y}_{1})}({x}_{2}-x)(y-{y}_{1})+\\ \;\;\;\;\;\;\;\;\;\;\;\;\;\; \frac{f\left({Q}_{22}\right)}{({x}_{2}-{x}_{1})({y}_{2}-{y}_{1})}(x-{x}_{1})(y-{y}_{1})\end{array} $ | (6) |
其中,
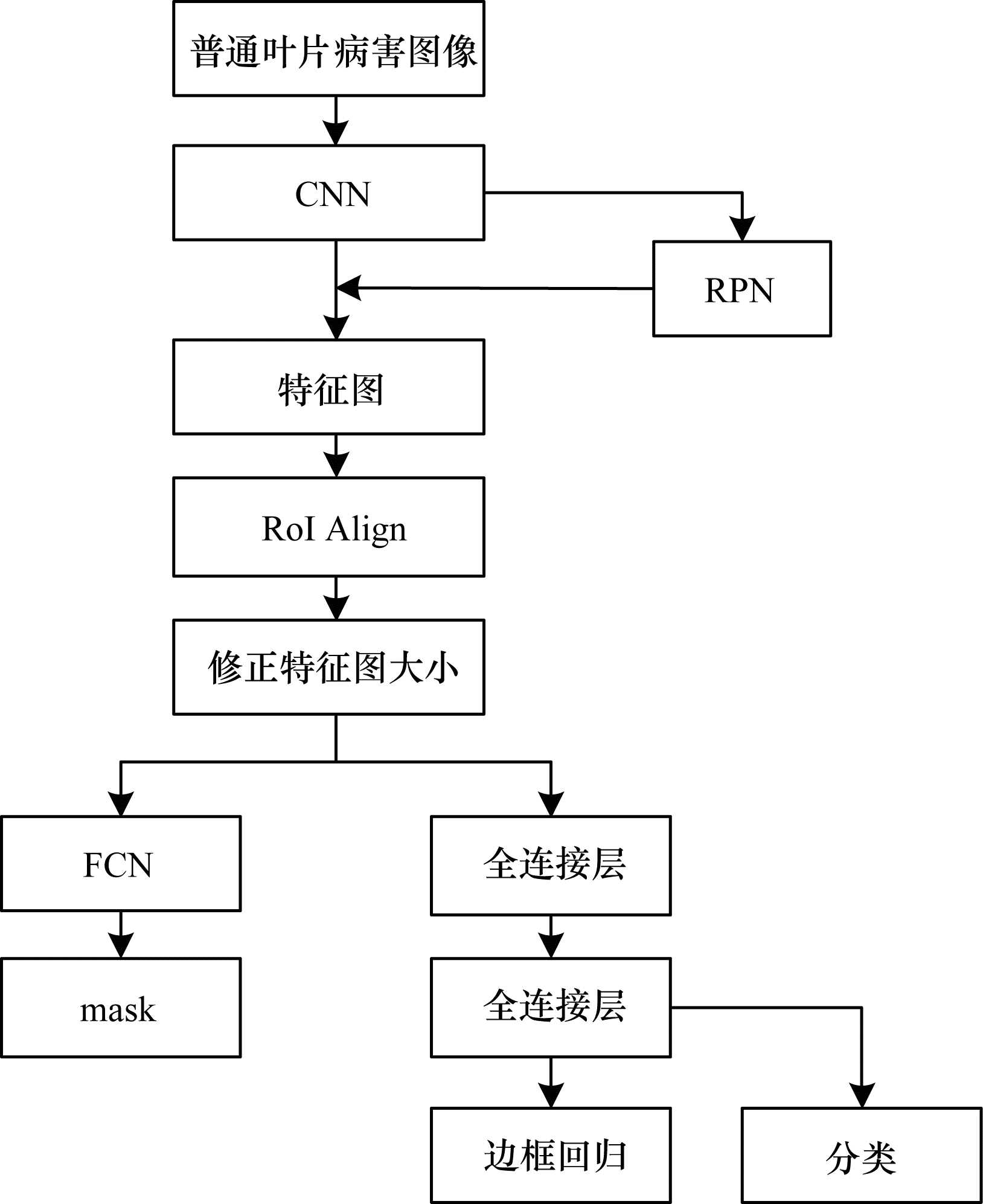
将图像缩放到统一大小后使用LabelMe[21]标注数据集,标注类别为葡萄叶片和背景区域。标注数据集后将葡萄叶片病害图像输入Mask R-CNN网络中进行检测,选取ResNet101作为Mask R-CNN的主干网络,将Microsoft COCO数据集上训练好的Mask R-CNN模型迁移至葡萄叶片病害数据集上进行训练,并对网络模型的参数值进行初始化。Mask R-CNN是由何凯明等人在原始Faster R-CNN[22]基础上增加全卷积网络(Fully Convolutional Networks,FCN)[23]而改进得到的,可以快速准确地进行目标检测和精准语义分割。Mask R-CNN网络结构如图 3所示,该框架可以用来完成目标检测、目标分类和像素级目标分割,且具有良好的扩展性和易用性。
|
Download:
|
| 图 3 Mask R-CNN网络结构 Fig. 3 Mask R-CNN network structure | |
Mask R-CNN的创新点在于采用RoI Align解决了像素偏差(Misalignment)问题。RoI Align通过遍历每个候选区域而将其分割成K×K个单元,采用双线性内插法计算每个单元中4个坐标的位置并进行最大池化。
在输入图像到CNN网络中获得对应的特征图后,从特征图中获得多个候选RoI,再将这些候选的RoI发送至RPN网络进行二值分类和边框回归,从而过滤掉一部分候选RoI,并对剩余的RoI进行RoI Align操作得到大小固定的特征图。将上述RoI通过全连接层进行分类,边框回归通过对每一个RoI进行FCN操作,从而生成mask。
经过Mask R-CNN处理后可得到葡萄叶片的mask图像,对图像的非叶片区域进行零像素填充,即保留葡萄叶片的像素值,其余部分填充零像素。本文提取的葡萄叶片病害图像如图 4所示。根据填充后的图像中叶片区域占比决定对叶片是否进行缩放,当占比值低于50%时,对mask图像再次使用双线性插值法将叶片区域恢复至原始大小,便于后续识别网络进行有效识别。

|
Download:
|
| 图 4 本文提取的葡萄叶片病害图像 Fig. 4 Grape leaf disease image extracted by this paper | |
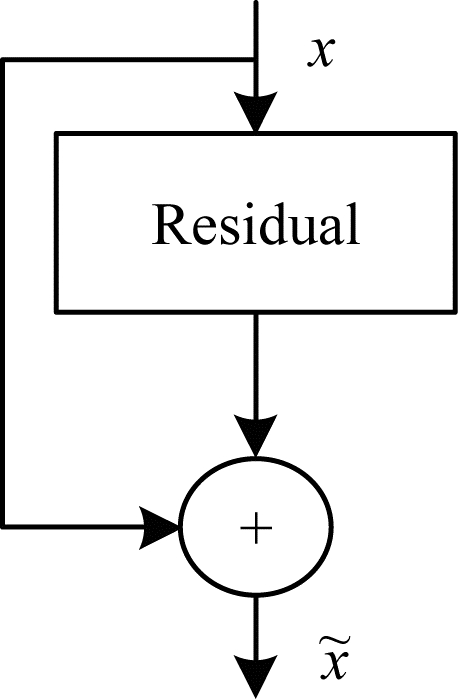
ResNet通常被广泛用于病害和虫害的识别[24-26],它通过增加网络的深度有效提升神经网络的特征提取性能,并采用残差学习的思想在一定程度上解决了信息传递时信息丢失、损耗和梯度消失与爆炸等网络退化问题。残差网络中通过直接添加输入信息到输出来保证原始信息的完整性。典型的残差块结构如图 5所示。
|
Download:
|
| 图 5 残差块结构 Fig. 5 Structure of residual block | |
葡萄叶片病害在不同时期具有不同的表征,比如黑痘病和褐斑病一般程度的葡萄叶片病斑较小且较为稀疏,多数低于26像素×28像素,而严重程度的病斑较大且较为密集,多数高于56像素×66像素,毛毡病的病斑区域较小且通常较为稀疏,多数低于19像素×12像素。由于固定尺度的卷积核在感知不同大小的病斑时效果不佳,因此本文将原始ResNet中conv1的7×7×64卷积改为多卷积组合结构,并采用数量依次为48、24和24的7×7、5×5和3×3不同尺度进行特征提取,利用1×1卷积降低通道数,将ResNet18的conv1设置成不同尺寸的卷积核组合的形式,以改善网络底层对不同特征的响应。多卷积组合结构如图 6所示,加入多卷积后的ResNet18与原始ResNet18网络结构对比如表 2所示。
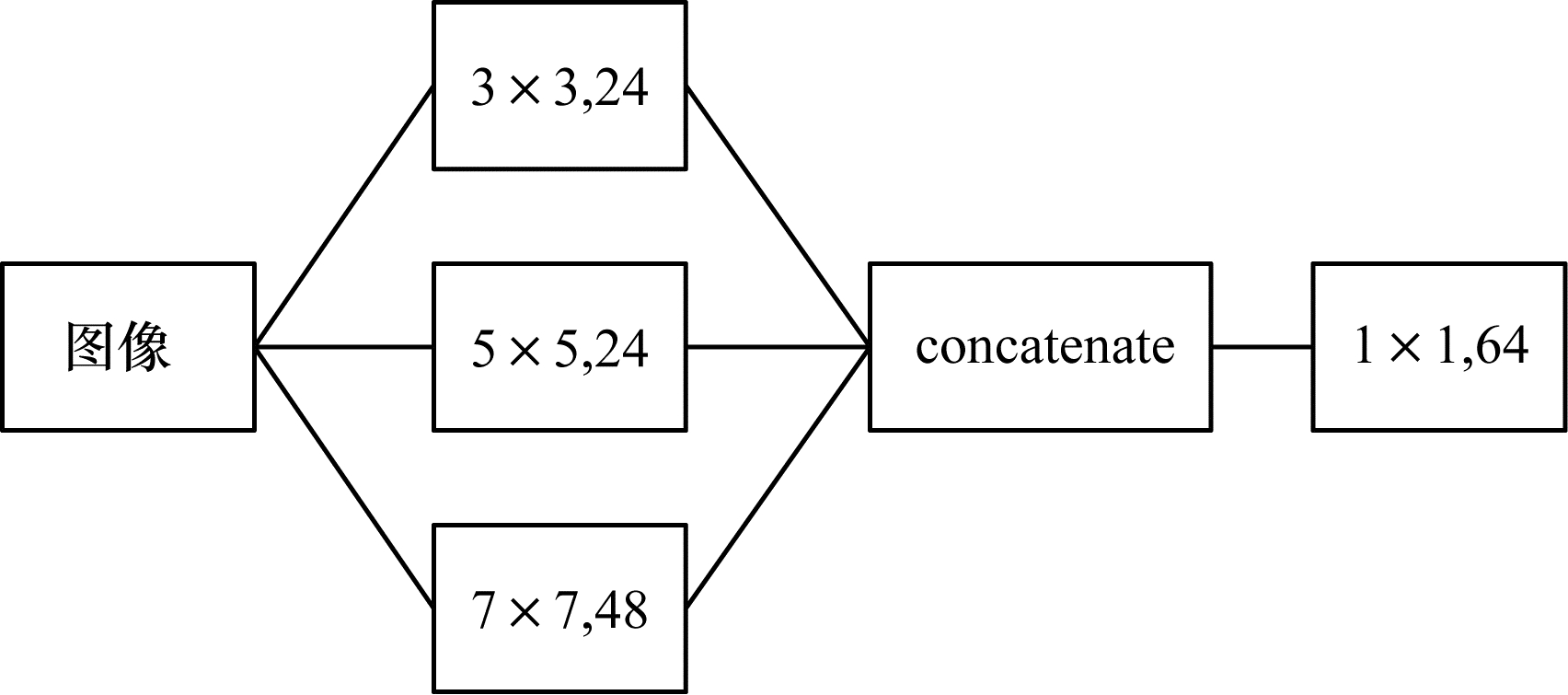
|
Download:
|
| 图 6 多卷积组合结构 Fig. 6 Multi-convolution combination structure | |

|
下载CSV 表 2 原始ResNet18和改进ResNet18结构对比 Table 2 Structure comparison between original ResNet18 and improved ResNet18 |
文献[19]从特征通道间的关系出发,提出SE(Squeeze-and-Excitation)模块。SE模块先对卷积得到的特征图进行挤压(Squeeze),即将每个二维特征通道变为一个实数从而得到通道级的全局特征,再对全局特征进行激励(Excitation)以学习各个通道间的关系,从而得到不同通道的权重,并与原始特征图相乘得到最终特征图。SE模块是在通道维度上加上注意力机制,该注意力机制可使得模型更加关注信息量最大的通道特征且抑制不重要的通道特征,即学习通道之间的相关性从而对不同通道进行权重分配,以帮助网络学习重要特征。基于SE模块注意力机制,可将SE模块与现有的网络结构进行融合[27]。
通过在ResNet18结构中加入SE模块,构成的新结构如图 7所示。网络中引入2个全连接层,用于限制模型复杂度、辅助增加模型的泛化能力,并在求和之前对残差部分进行特征重定向。SE模块使得神经网络根据重要度提高对网络有用的特征并抑制用处较小的特征。
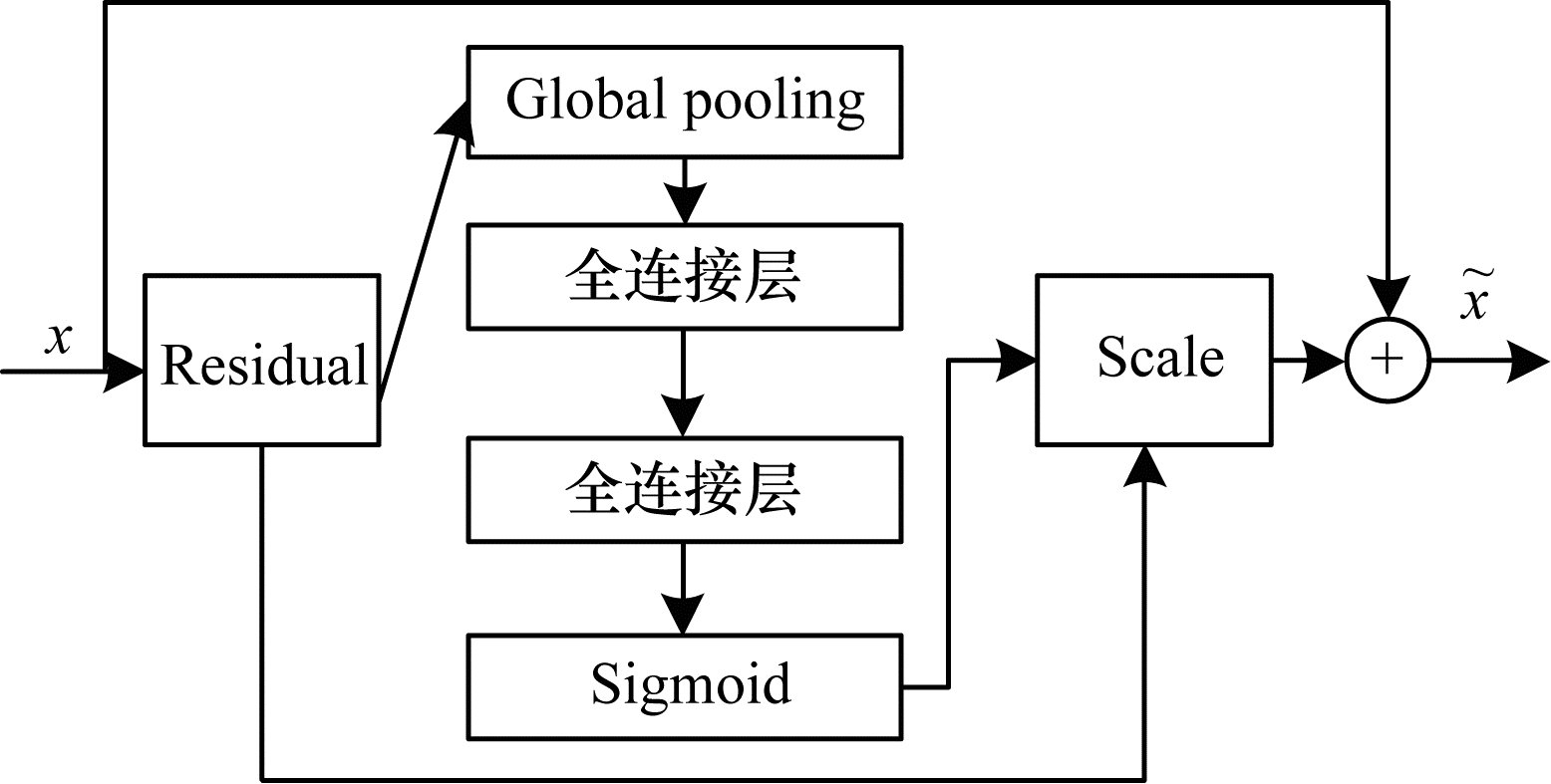
|
Download:
|
| 图 7 加入SENet后的ResNet结构 Fig. 7 ResNet structure after adding SENet | |
实验环境配置如表 3所示,Mask R-CNN的主干网络选用ResNet101,先训练20 epoch的head layers,再训练40 epoch的all layers。Multi-Scale ResNet模型选用Momentum优化器,且分别训练100 epoch、70 epoch与90 epoch时,学习率初始值分别选取为0.01、0.001和0.000 1,动量值均为0.9,batch设置为64,选用识别准确率作为评价指标。

|
下载CSV 表 3 实验环境配置 Table 3 Experimental environment configuration |
为验证Multi-Scale ResNet的有效性,实验对SVM、Inception-ResNet、ResNeXt-50和文献[10]中的UnitedModel这4种方法的识别准确率进行对比分析,结果如表 4所示。其中,SVM方法的主要过程是先使用Mask R-CNN提取葡萄叶片,再提取各病斑的HSV颜色特征和HOG纹理特征,并输入SVM中获取结果。从表 4可以看出,SVM对葡萄叶片病害的识别准确率较低,这是因为它在识别过程中需要人工设计特征进行特征提取,而颜色和纹理特征比较有限,同种病害的一般程度和严重程度在颜色和纹理特征方面较为相似,黑痘病与褐斑病在葡萄叶片的病斑表象比较相似,毛毡病病斑颜色与叶片正常部位相似,因此不能较好的识别,且对于不同的病害需要提取不同的特征,不具有广泛的适用性。Inception-ResNet和ResNeXt-50的平均识别准确率分别为87.00%和87.68%,这2种模型对褐斑病严重程度和黑痘病一般程度的识别准确率较低。Inception-ResNet通过使用1
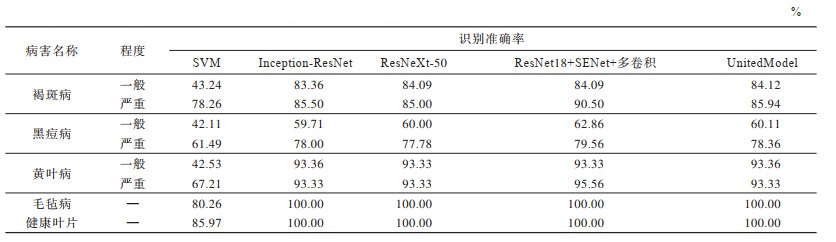
|
下载CSV 表 4 不同方法的识别准确率对比 Table 4 Comparison of recognition accuracy of different methods |
为确定ResNet的层数,实验对ResNet18、ResNet50和ResNet101这3种结构的拟合能力进行对比,结果如表 5所示。
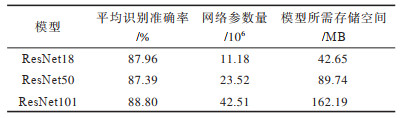
|
下载CSV 表 5 3种结构的拟合能力对比 Table 5 Comparison of fitting ability of three structures |
从表 5可以看出,ResNet101的平均识别准确率最高可达88.80%,ResNet50比ResNet18的平均识别准确率低,这是因为ResNet50对褐斑病和黑痘病一般程度的识别准确率虽然较高,但是对其他程度的病害的识别准确率均有所下降,最终导致识别准确率下降。ResNet18在平均识别准确率比ResNet101低0.84个百分点的情况下,网络参数减少了31.33
为确定多卷积核组合时不同卷积核个数对模型的影响,针对(7×7,64),
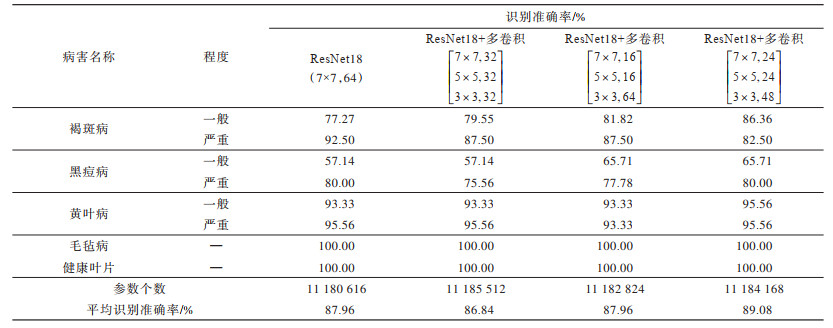
|
下载CSV 表 6 卷积核个数对本文模型识别准确率的影响 Table 6 Effect of convolution kernel number on recognition accuracy of the proposed model |
为增强实验结果的准确性并验证改进点对模型的影响,针对多种卷积核组合和SE模块这2个改进点,将改进点与ResNet18模型分别组合为以下4组模型:1)原始ResNet18;2)ResNet18+多卷积;3)ResNet18+SENet;4)ResNet18+SENet+多卷积。实验对上述4种模型的识别准确率和平均识别准确率进行对比,结果如表 7所示。
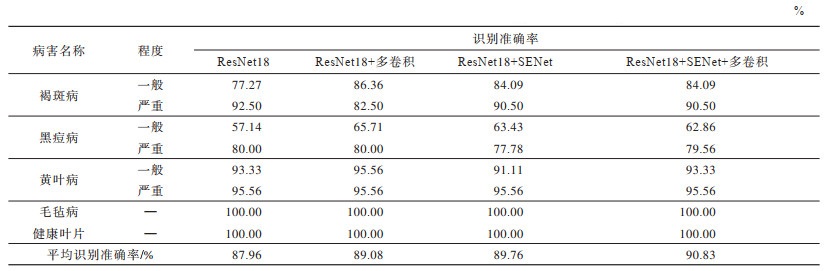
|
下载CSV 表 7 多卷积与SENet对识别准确率的影响 Table 7 Effect of multi-scale and SENet on recognition accuracy |
从表 7可以看出,加入多卷积的ResNet18对褐斑病严重程度的识别准确率相比ResNet18下降了10个百分点,这是因为褐斑病严重程度的葡萄叶片中呈现出连续的大片病斑区域,多卷积核中对于大卷积核的个数设置的比原始卷积少,因此识别准确率下降。加入SE模块的ResNet18在褐斑病一般程度、黑痘病一般程度上平均识别准确率相比原始ResNet分别提升了6.82和6.29个百分点,但是对黑痘病严重程度和黄叶病一般程度的识别准确率均下降了2.22个百分点,这是因为SENet注意力机制能够自动获取各特征通道的重要性,使得网络学习到更加重要的特征,从而在网络学习特征时更加均衡,这种均衡导致对褐斑病一般程度和黑痘病一般程度的识别准确率提升,而对于褐斑病和黑痘病严重程度的识别准确率有所下降。同时加入SE模块和多卷积结构在平均识别准确率上达到了90.83%,相比原始ResNet18提高了2.87个百分点,弥补了单独加入SE模块和多卷积结构的不足,解决了仅加入多卷积核组合或仅加入SE模块造成部分病害识别准确率下降的问题。
4 结束语为提高对不同程度葡萄叶片病害的识别准确率,本文提出一种基于Multi-Scale ResNet的葡萄叶片病害识别方法。该方法使用Mask R-CNN提取葡萄叶片,以减少复杂背景区域的影响,并把提取的病害图像输入改进的ResNet18网络中,通过将ResNet18的conv1层改为多卷积核组合的方式改变网络对不同特征的响应,同时在ResNet18中加入SENet模块以提高网络特征提取能力。实验结果表明,本文方法相比传统SVM方法的适用性更强,且平均识别准确率达到90.83%。下一步将结合低照度增强算法研究不同照度对病害识别准确率的影响,以进一步提高模型的鲁棒性。
| [1] |
PUJARI J D, YAKKUNDIMATH R, BYADGI A S. SVM and ANN based classification of plant diseases using feature reduction technique[J]. International Journal of Interactive Multimedia and Artificial Intelligence, 2016, 3: 6-14. |
| [2] |
WANG Dan, CHAI Xiujuan. Application of machine learning in plant diseases recognition[J]. Journal of Chinese Agricultural Mechanization, 2019, 40(9): 171-180. (in Chinese) 王聃, 柴秀娟. 机器学习在植物病害识别研究中的应用[J]. 中国农机化学报, 2019, 40(9): 171-180. |
| [3] |
LIU Bin, ZHANG Yun, HE Dongjian, et al. Identification of apple leaf diseases based on deep convolutional neural networks[J]. Symmetry, 2018, 10(1): 11-18. |
| [4] |
LI Xinxing, ZHU Chenguang, BAI Xuebing, et al. Recognition method of cucumber leaves diseases based on visual spectrum and support vector machine[J]. Spectroscopy and Spectral Analysis, 2019, 39(7): 2250-2256. (in Chinese) 李鑫星, 朱晨光, 白雪冰, 等. 基于可见光谱和支持向量机的黄瓜叶部病害识别方法研究[J]. 光谱学与光谱分析, 2019, 39(7): 2250-2256. |
| [5] |
HUANG Shuangping, SUN Chao, QI Long, et al. Rice panicle blast identification method based on deep convolution neural network[J]. Transactions of the Chinese Society of Agricultural Engineering, 2017, 33(20): 169-176. (in Chinese) 黄双萍, 孙超, 齐龙, 等. 基于深度卷积神经网络的水稻穗瘟病检测方法[J]. 农业工程学报, 2017, 33(20): 169-176. DOI:10.11975/j.issn.1002-6819.2017.20.021 |
| [6] |
SUN Jun, TAN Wenjun, MAO Hanping, et al. Recognition of multiple plant leaf diseases based on improved convolutional neural network[J]. Transactions of the Chinese Society of Agricultural Engineering, 2017, 33(19): 209-215. (in Chinese) 孙俊, 谭文军, 毛罕平, 等. 基于改进卷积神经网络的多种植物叶片病害识别[J]. 农业工程学报, 2017, 33(19): 209-215. DOI:10.11975/j.issn.1002-6819.2017.19.027 |
| [7] |
LONG Mansheng, OUYANG Chunjuan, LIU Huan, et al. Image recognition of Camellia oleifera diseases based on convolutional neural network & transfer learning[J]. Transactions of the Chinese Society of Agricultural Engineering, 2018, 34(18): 194-201. (in Chinese) 龙满生, 欧阳春娟, 刘欢, 等. 基于卷积神经网络与迁移学习的油茶病害图像识别[J]. 农业工程学报, 2018, 34(18): 194-201. DOI:10.11975/j.issn.1002-6819.2018.18.024 |
| [8] |
WANG Guan, SUN Yu, WANG Jianxin. Automatic image-based plant disease severity estimation using deep learning[J]. Computational Intelligence and Neuroscience, 2017, 2017: 1-8. |
| [9] |
AGRAWAL N, SINGHAI J, AGARWAL D K. Grape leaf disease detection and classification using multi-class support vector machine[C]//Proceedings of 2017 International Conference on Recent Innovations in Signal processing and Embedded Systems. Berlin, Germany: Springer, 2017: 238-244.
|
| [10] |
JI Miaomiao, ZHANG Lei, WU Qiufeng. Automatic grape leaf diseases identification via UnitedModel based on multiple convolutional neural networks[J]. Information Processing in Agriculture, 2020, 7(3): 418-426. DOI:10.1016/j.inpa.2019.10.003 |
| [11] |
QIAO Hong, FENG Quan, ZHANG Rui, et al. Dynamic monitoring of grape leaf disease based on sequential images tracking[J]. Transactions of the Chinese Society of Agricultural Engineering, 2018, 34(17): 167-175. (in Chinese) 乔虹, 冯全, 张芮, 等. 基于时序图像跟踪的葡萄叶片病害动态监测[J]. 农业工程学报, 2018, 34(17): 167-175. DOI:10.11975/j.issn.1002-6819.2018.17.022 |
| [12] |
LIU Yang, FENG Quan, WANG Shuzhi. Plant disease identification method based on lightweight CNN and mobile application[J]. Transactions of the Chinese Society of Agricultural Engineering, 2019, 35(17): 194-204. (in Chinese) 刘洋, 冯全, 王书志. 基于轻量级CNN的植物病害识别方法及移动端应用[J]. 农业工程学报, 2019, 35(17): 194-204. DOI:10.11975/j.issn.1002-6819.2019.17.024 |
| [13] |
WU Ang, ZHU Juanhua, HE Yujing. Computer vision method applied for detecting diseases in grape leaf system[C]//Proceedings of Cognitive Internet of Things: Frameworks, Tools and Applications. Berlin, Germany: Springer, 2018: 367-376.
|
| [14] |
ADEEL A, KHAN M A, SHARIF M, et al. Diagnosis and recognition of grape leaf diseases: an automated system based on a novel saliency approach and canonical correlation analysis based multiple features fusion[J]. Sustainable Computing: Informatics and Systems, 2019, 24: 126-136. |
| [15] |
JAISAKTHI S M, MIRUNALINI P, THENMOZHI D, et al. Grape leaf disease identification using machine learning techniques[C]//Proceedings of International Conference on Computational Intelligence in Data Science. Berlin, Germany: Springer, 2019: 1-6.
|
| [16] |
ZHU Juanhua, WU Ang, WANG Xiushan, et al. Identification of grape diseases using image analysis and BP neural networks[J]. Multimedia Tools and Applications, 2020, 79(21/22): 14539-14551. DOI:10.1007/s11042-018-7092-0 |
| [17] |
HE Kaiming, ZHANG Xiangyu, REN Shaoqing, et al. Deep residual learning for image recognition[C]//Proceedings of 2016 IEEE Conference on Computer Vision and Pattern Recognition, Washington D.C., USA: IEEE Press, 2016: 770-778.
|
| [18] |
GUO Xiaoqing, FAN Taojie, SHU Xin. Tomato leaf diseases recognition based on improved Multi-Scale AlexNet[J]. Transactions of the Chinese Society of Agricultural Engineering, 2019, 35(13): 162-169. (in Chinese) 郭小清, 范涛杰, 舒欣. 基于改进Multi-Scale AlexNet的番茄叶部病害图像识别[J]. 农业工程学报, 2019, 35(13): 162-169. DOI:10.11975/j.issn.1002-6819.2019.13.018 |
| [19] |
HU J, LI S, ALBANIE S, et al. Squeeze-and-excitation networks[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 42(8): 2011-2023. |
| [20] |
HE K M, GKIOXARI G, DOLLAR P, et al. Mask R-CNN[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2017: 2961-2969.
|
| [21] |
RUSSELL B C, TORRALBA A, MURPHY K P, et al. LabelMe: a database and Web-based tool for image annotation[J]. International Journal of Computer Vision, 2008, 77(1/2/3): 157-173. |
| [22] |
REN S Q, HE K M, GIRSHICK R, et al. Faster R-CNN: towards real-time object detection with region proposal networks[J]. IEEE Transactions on Pattern Analysis & Machine Intelligence, 2015, 39(6): 1137-1149. |
| [23] |
JONATHAN L, EVAN S, TREVOR D. Fully convolutional networks for semantic segmentation[C]//Proceedings of 2015 IEEE Conference on Computer Vision and Pattern Recognition. Washington D.C., USA: IEEE Press, 2015: 1138-1146.
|
| [24] |
CHEN Juan, CHEN Liangyong, WANG Shengsheng, et al. Pest image recognition of garden based on improved residual network[J]. Transactions of the Chinese Society for Agricultural Machinery, 2019, 50(5): 187-195. (in Chinese) 陈娟, 陈良勇, 王生生, 等. 基于改进残差网络的园林害虫图像识别[J]. 农业机械学报, 2019, 50(5): 187-195. |
| [25] |
WANG Dandan, HE Dongjian. Recognition of apple targets before fruits thinning by robot based on R-FCN deep convolution neural network[J]. Transactions of the Chinese Society of Agricultural Engineering, 2019, 35(3): 156-163. (in Chinese) 王丹丹, 何东健. 基于R-FCN深度卷积神经网络的机器人疏果前苹果目标的识别[J]. 农业工程学报, 2019, 35(3): 156-163. |
| [26] |
ZENG Weihui, LI Miao, LI Zeng, et al. High-order residual and parameter-sharing feedback convolutional neural network for crop disease recognition[J]. Acta Electronica Sinica, 2019, 47(9): 1979-1986. (in Chinese) 曾伟辉, 李淼, 李增, 等. 基于高阶残差和参数共享反馈卷积神经网络的农作物病害识别[J]. 电子学报, 2019, 47(9): 1979-1986. DOI:10.3969/j.issn.0372-2112.2019.09.023 |
| [27] |
LIU Xueping, LI Yuqian, LIU Li, et al. Improved YOLOV3 target recognition algorithm with embedded SENet structure[J]. Computer Engineering, 2019, 45(11): 243-248. (in Chinese) 刘学平, 李玙乾, 刘励, 等. 嵌入SENet结构的改进YOLOV3目标识别算法[J]. 计算机工程, 2019, 45(11): 243-248. |
 2021, Vol. 47
2021, Vol. 47

 ,
,