2. 德州市人民医院 肿瘤科, 山东 德州 253000;
3. 厦门理工学院 计算机与信息工程学院, 福建 厦门 361024
2. Department of Oncology, Dezhou People's Hospital, Dezhou, Shandong 253000, China;
3. College of Computer and Information Engineering, Xiamen University of Technology, Xiamen, Fujian 361024, China
开放科学(资源服务)标志码(OSID):
我国是世界上食管癌病死率较高的国家,其发病率在各类肿瘤中居第3位,病死率居第4位[1-2]。食管癌在我国不同地区的发病率有明显差异,受某些诱导因素的影响,有高发病率地区和低发病率地区。因此,对高发病区域进行食管癌肿瘤的筛查诊断是非常有效的防控手段[3]。
食管癌的诊治是医生借助电子计算机断层扫描(CT)、核磁共振成像(MRI)等技术对生成的医学影像进行分析。由于人工阅片的工作量非常庞大,因此医生可能会因长时间阅片产生疲劳感,影响对病灶的判断,增大漏诊、误诊的风险。此外,疾病的检出率很大程度上依赖于影像科医生的主观经验,易受本人生理、心理等状态的影响,存在一定的诊断偏差,从而影响诊断结果的准确性[4-6]。因此,根据食管癌医学影像进行肿瘤检测和准确分割,是进行病灶切除手术的前提,同时深度学习自动化处理方式也对辅助医生诊断治疗有重要意义。
近年来,基于深度学习的医学影像分析在医疗领域取得了一定的成果,在自动识别可疑病灶、对病灶进行量化诊断等方面取得新进展[6]。但是,受数据集大小以及数据标注准确性等因素的影响,现有研究往往将肺癌、乳腺癌等常见癌症作为检测目标。然而,采用深度学习诊断肿瘤的方式并不具有跨病种的泛化能力,即对某一疾病诊断效果好的深度学习网络通常不适用于其他疾病。因此,研究人员尝试对传统肿瘤检测网络进行改进,以适用于诊断新的疾病。
本文设计基于U-Net的图像分割网络,以精准分割食管癌肿瘤。通过构建Conv_concat模块,并引入2个跳跃连接,增强图像特征信息的传递性,在Conv_concat模块的每一个卷积层后都增加批量归一化(Batch Normalization,BN)层[7],提升网络训练稳定性。在此基础上,结合BCEWithLogits和Dice损失函数的优点,改进网络的损失函数,从而提高训练效率和分割准确率。
1 相关工作 1.1 图像分割网络图像分割技术是能够精确地把肿瘤从影像中分割出。早期,医学图像分割方法主要有基于边缘的图像分割[8]、基于区域的图像分割等[9]。之后,研究人员提出基于图论的图像分割方法,将目标图像映射为带权的无向图,进而把图像分割问题转换为图的最小割(MIN-CUT)问题,因此只需保证子图间的相似度最小,而内部的相似度最大即可[10-12]。
为了对存在不确定性或模糊性的图像进行准确分割,研究人员将机器学习中的聚类方法应用于图像分割问题,产生了一系列基于像素聚类的分割算法,例如,k均值聚类算法[13]、谱聚类[14]、均值漂移[15]以及用于超像素聚类的简单线性迭代聚类算法[16]等。这种特征空间聚类法在一定程度上满足了研究人员对图像分割的需求。文献[17]提出一种基于k均值聚类的图像分割算法,并进行脑部核磁共振图像的分割,能较准确地分割出脑部核磁共振成像中的灰质和白质,噪声和灰度不均匀的图像上也能在较短的时间内得到更准确的结果。文献[18]提出使用核函数将原始数据从低维空间映射到高维空间,并在高维空间中利用图割理论对CT图像的肝区肿瘤进行分割,提取疑似肿瘤区域,以解决传统图割模型依赖人机交互且对复杂区域建模困难等问题。但是,对于图像内部结构复杂的物体,仅通过图像像素点的颜色、亮度、纹理等低层次的视觉特征进行图像分割,实际效果往往与预期相差较大。
为了解决这个问题,研究人员结合图像中的高层语义信息,使用语义分割来辅助图像分割。为实现图像像素级别的分割,研究人员对图像的每个像素进行分类,使得每个像素都具有高阶的语义信息,从而提高分割的准确率。LONG[19]等提出全卷积网络(FCN),将目标识别网络中的全连接层替换成卷积层和池化层,从而把分类网络转换成能够进行图像分割的全卷积网络,不仅实现了像素级的稠密估计目的,而且能够进行端到端的网络训练。TONG[20]提出一种新的分割模型,将形状表示模型集成到FCN中以提升CT图像器官分割的精度,但是FCN本身具有一定的局限性,例如感受野太小,无法获取全局信息,导致网络学习性能受到限制。
针对这些问题,研究人员在FCN编码解码结构的基础上,对其局限性做了相应的改进。RONNEBERGER[21]等提出U-Net网络架构对医学图像进行分割,该网络把编码解码2个过程设计为1个对称的U型结构,并在不同层之间增加跳跃连接[22]操作,实现了高维特征与低维特征的融合,使得像素定位更加准确,分割精度更高。BROSCH[23]等在U-Net网络下采样的第一层和上采样的最后一层之间加入跳跃连接操作,在一定程度上提高了分割精度,在分割脑部MRI中的脑白质病灶中取得了较优的效果。文献[24]使用内卷U-Net对肺部CT数据进行分割。内卷U-Net使用内卷积代替传统的卷积操作,同时该方法使用注意力机制学习图像长范围的依赖关系,以提高图像分割精度。文献[25]针对食管癌的X射线,通过U-Net结合图像插值算法处理U-Net分割轮廓图小部分不连续的问题。
深度图像分割网络一般都是在FCN的编码解码结构基础上构建的。针对在FCN的池化和上采样过程中信息丢失的问题,CHEN等在DeepLab系列[26-28]网络中使用空洞卷积,并提出ASPP(Atrous Spatial Pyramid Pooling)模块,使其在不增加参数量的前提下扩大特征图的视野,增强对感受野和分辨率的控制,有利于提取全局信息,提高分割准确率。LIU等[29]基于池化的全局特征,利用ParseNet实现全局特征与局部特征融合的图像分割,从而提高分割精度。BADRINARAYANAN等[30]提出SegNet网络,通过引入池化层索引来提高分割精度。SegNet和FCN的原理相似,不同之处在于解码器使用从编码器传输的对应位置最大池化层,对其输入进行非线性上采样,从而使得上采样不需要学习即可生成稀疏特征映射,通过可训练的卷积核进行卷积操作,以生成密集的特征图,最后使用Softmax进行像素级别分类。ROMER等[31]提出轻量级的语义分割网络ERFNet,ERFNet一方面使用了1×1的卷积核减少网络计算参数量,从而加快网络速度,另一方面采用与ResNet[32]主干网络相同的残差连接方式提升模型精度。
1.2 U-Net网络在医学图像分割领域中,U-Net网络具有较高的训练效率以及能够适用少量训练样本的特征,备受研究人员的关注。U-Net的设计主要针对医学影像的分割,其网络结构与FCN类似。在FCN编码解码结构的基础上,U-Net网络将原来的直线型网络结构变成了一个对称的U型结构,只需要较小的数据集就可以完成精准的图像分割。在U-Net网络中还增加跳跃连接结构,实现了图像的低层信息与高层信息在特征提取过程中的融合,从而提高分割精度。U-Net包括编码结构、解码结构和跳跃连接结构。
U-Net的编码结构实现了图像特征的提取,该结构由2种操作重复叠加组成:1)卷积操作,由2个连续的卷积块构成,每个卷积层的后面都要经过修正线性单元ReLU[33]激活层,用于提高网络训练的稳定性;2)最大池化操作,能够降低空间分辨率,提取抽象的高维特征,以提升网络的鲁棒性。U-Net的解码结构与编码结构相对应,也包含卷积操作和上采样操作,采用ConvTranspose[23]方法进行上采样,通过2×2的反卷积逐步恢复图像的分辨率,最终输出分割结果的热力图。
U-Net通过在编码与解码之间增加跳跃连接,对FCN进行改进,将编码结构每一层产生的特征图融合到解码结构对应层的上采样过程中,实现低层信息与高层信息的融合。在融合过程中,编码结构的卷积操作会导致边界像素丢失。因此,U-Net融合2个特征图的大小并不相同,对编码结构的特征图进行裁剪,再进行拼接。由于每次融合前都要先进行裁剪,因此最后输出图像的大小比原图小很多。但是由于增加了跳跃连接操作,因此图像的分割精度得到显著提高。
2 本文方法本文以U-Net网络作为基础网络,通过改进卷积块、损失函数,加入早停机制等方法构建适用于食管癌肿瘤分割的Concat-UNet网络。
与U-Net网络类似,Concat-UNet网络也包含编码、解码和跳跃连接3种主要结构。Concat-UNet网络结构如图 1所示。编码结构包含Conv_concat模块和最大池化层,Conv_concat可以有效融合特征,最大池化层通过保留有效特征,丢弃不明显的特征来实现下采样。解码结构包含Conv_concat模块和上采样,使用与U-Net相同的上采样方法恢复图片的分辨率,弥补在池化过程中丢失的细节信息,1×1卷积会根据分类需求对图片中的像素进行分类,以得到最终的结果。
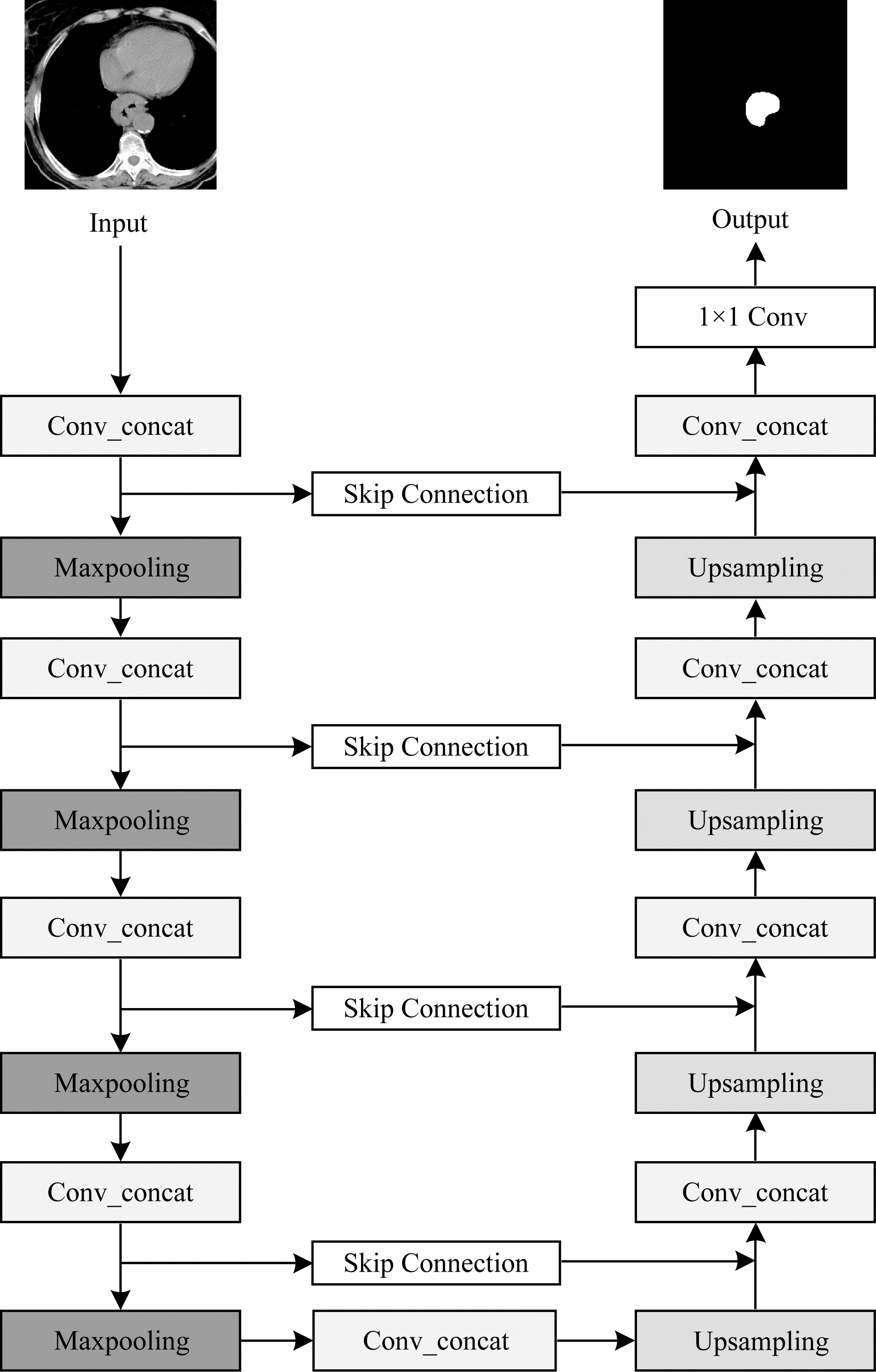
|
Download:
|
| 图 1 Concat-UNet的网络结构 Fig. 1 Structure of Concat-UNet network | |
Concat-UNet通过改进U-Net的卷积块,构建新的Conv_concat模块。改进后的卷积Conv_concat模块结构如图 2所示。U-Net网络中的卷积块结构如图 3所示。
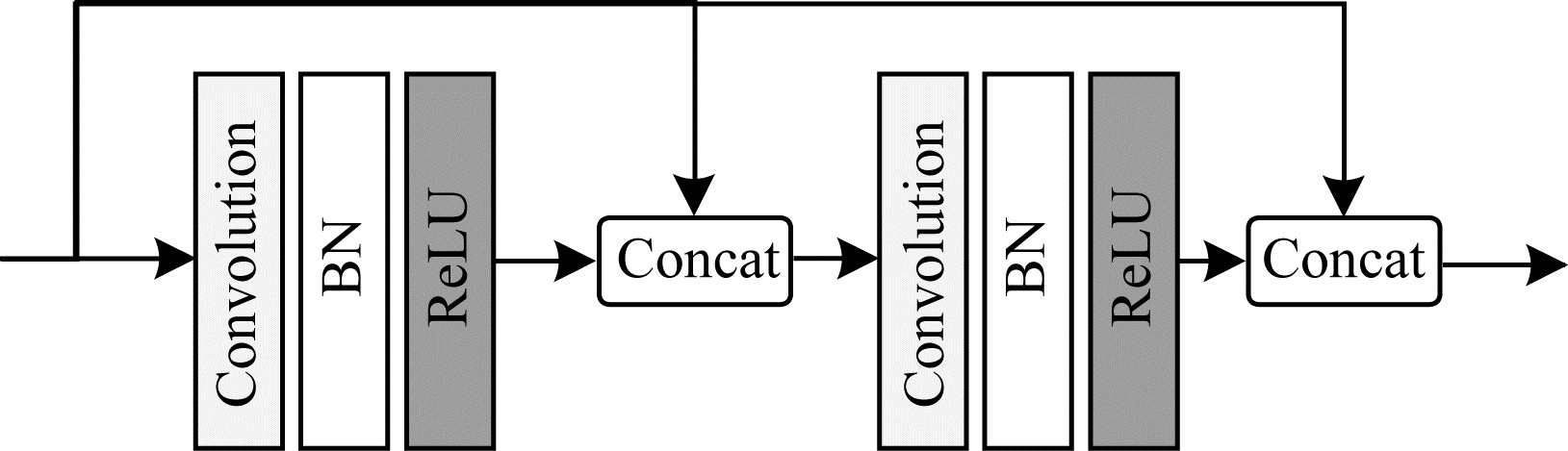
|
Download:
|
| 图 2 改进的Conv_concat模块结构 Fig. 2 Structure of improved Conv_concat module | |
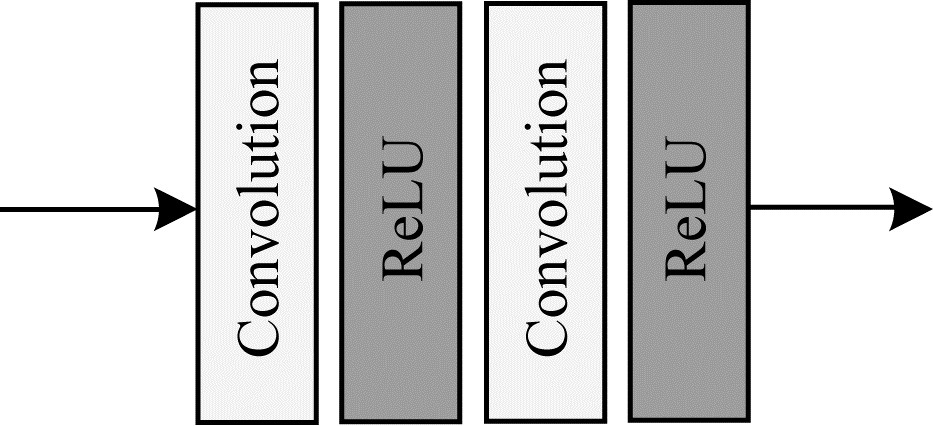
|
Download:
|
| 图 3 U-Net网络中的卷积块结构 Fig. 3 Structure of convolution block in U-Net network | |
从图 2和图 3可以看出,相较于原始U-Net网络的卷积块,Conv_concat模块做了2个方面的改进:1)将每次卷积产生的特征图与最初输入Conv_concat模块的特征图进行融合,并将融合结果作为下一次卷积的输入,这种改进方法增强了特征的传递性,在训练过程减少图像特征损失,从而提高分割精度,但是卷积操作会损失图像边缘信息,导致卷积前后图像大小不一致,因此为了使Conv_concat模块中卷积前后的特征图相融合,在卷积前先对特征图进行边缘填充,以保证卷积前后特征图分辨率的一致性,从而解决U-Net最终输出图片比原图片小的问题;2)Conv_concat模块在每一个卷积层后面都增加了一个批量归一化层,BN的主要作用是把每批次中的数据正态化,从而减少网络对初始化参数的依赖。对于任意输入的
为了避免网络优化受特征分布的影响,网络还需要对正态化后的数据
| $ {y}_{i}={\gamma }_{i}\cdot {x}_{i}+{\beta }_{i} $ | (1) |
其中:
为提高网络的泛化能力,本文引入早停机制,通过找出并保存训练过程中最优的网络参数,无需反复训练,从而避免网络训练过程中出现过拟合现象。
早停机制的示意图如图 4所示,当Concat-UNet网络在训练集上的得分逐渐增大时,在验证集上的得分反而降低,说明该网络有发生过拟合的趋势。当Concat-UNet网络连续P次(P为耐心值,即已经等待的次数)出现此种情况,则应引发早停机制。若当前的得分
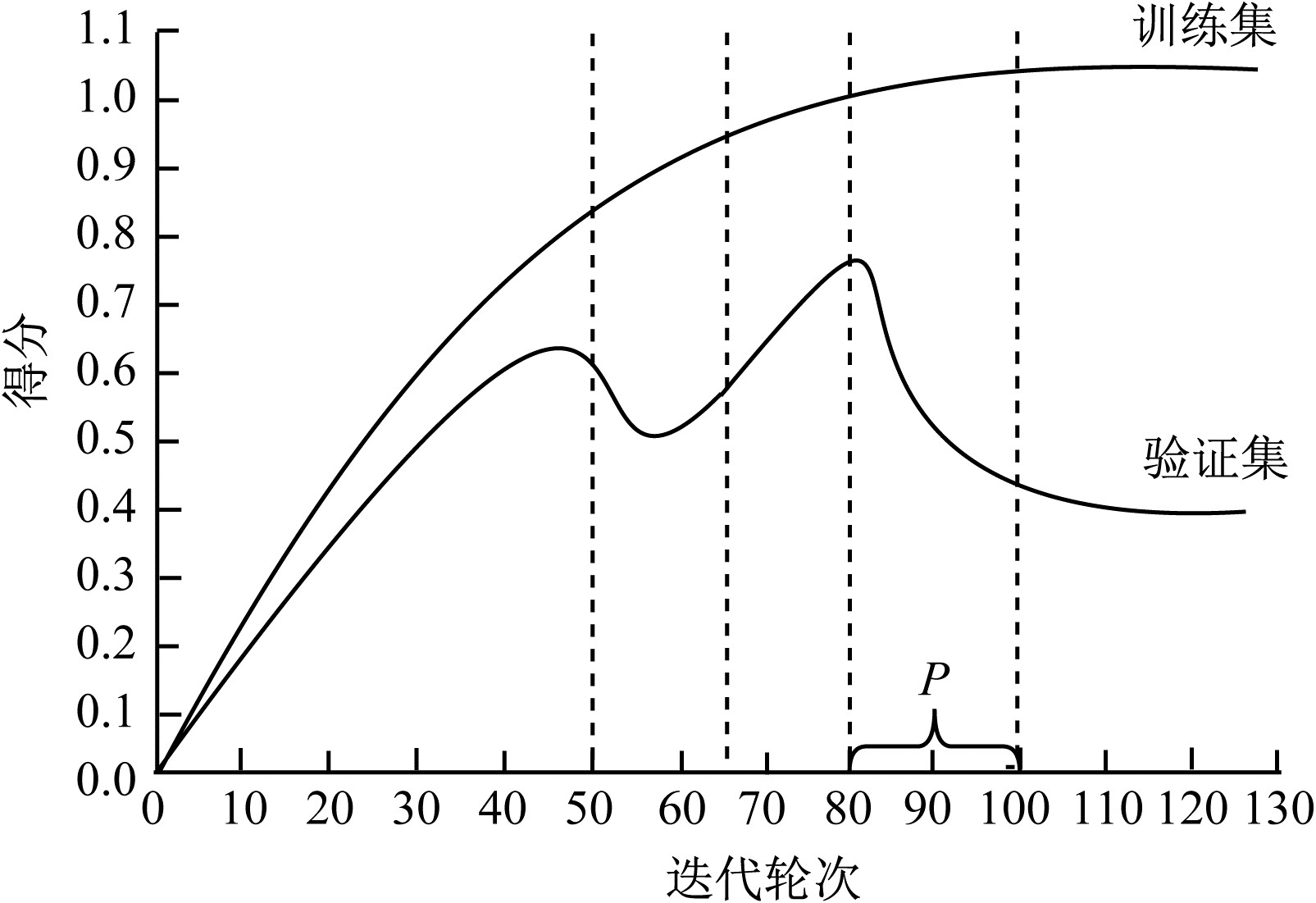
|
Download:
|
| 图 4 早停机制示意图 Fig. 4 Schematic diagram of early stopping mechanism | |
在医学图像分割领域中,常用的损失函数有二值交叉熵(Binary Cross Entropy,BCE)、BCEWithLogits、Dice等,这些损失函数都各有优点和局限性。本文结合BCEWithLogits和Dice损失函数的优点,设计新的的损失函数。
2.2.1 BCEWithLogits损失函数传统方法使用BCE损失函数来计算二分类交叉熵,要求样本必须在[0, 1]之间,若使用Sigmoid函数来实现,易导致计算结果不稳定,并且存在梯度消失的问题。针对该问题,本文使用BCEWithLogits损失函数,将Sigmoid函数和BCE损失函数集合到一个类中,使计算结果更加稳定,有利于梯度的稳定回传,避免出现梯度消失的问题。BCEWithLogits损失函数如式(2)所示:
| $ \begin{array}{l}{L}_{\mathrm{B}\mathrm{C}\mathrm{E}\mathrm{W}\mathrm{i}\mathrm{t}\mathrm{h}\mathrm{L}\mathrm{o}\mathrm{g}\mathrm{i}\mathrm{t}\mathrm{s}}=\\ -\sum\limits_{i=1}^{n}{y}_{i}lo{g}_{a}\left(\sigma \right({x}_{i}\left)\right)+(1-{y}_{i})\mathrm{l}\mathrm{o}{\mathrm{g}}_{\mathrm{a}}(1-\sigma ({x}_{i}\left)\right)\end{array} $ | (2) |
其中:
该损失函数会对每个类别进行平等的评估,不利于类别不平衡分割网络的优化。前景像素(肿瘤区域)远小于背景像素(非肿瘤区域),因此背景像素会在损失函数中占主导地位,使该网络偏向优化背景。
2.2.2 Dice损失函数Dice系数表征了2个样本重叠部分的情况,取值范围为[0, 1],取值为1表示完全重叠。Dice系数如式(3)所示:
| $ \mathrm{D}\mathrm{i}\mathrm{c}\mathrm{e}({G}_{\mathrm{G}\mathrm{T}}, {P}_{\mathrm{P}\mathrm{R}})=\frac{2\times |{G}_{\mathrm{G}\mathrm{T}}\bigcap {P}_{\mathrm{P}\mathrm{R}}|}{\left|{G}_{\mathrm{G}\mathrm{T}}\right|+\left|{P}_{\mathrm{P}\mathrm{R}}\right|} $ | (3) |
其中:
根据Dice系数,可求得Dice损失函数,如式(4)所示:
| $ {L}_{\mathrm{D}\mathrm{i}\mathrm{c}\mathrm{e}}=1-\frac{2\times \sum\limits_{\mathrm{p}\mathrm{i}\mathrm{x}\mathrm{e}\mathrm{l}\mathrm{s}}{G}_{GT}\cdot {P}_{PR}}{\sum\limits_{\mathrm{p}\mathrm{i}\mathrm{x}\mathrm{e}\mathrm{l}\mathrm{s}}{G}_{\mathrm{G}\mathrm{T}}^{2}+\sum\limits_{\mathrm{p}\mathrm{i}\mathrm{x}\mathrm{e}\mathrm{l}\mathrm{s}}{P}_{\mathrm{P}\mathrm{R}}^{2}} $ | (4) |
Dice损失函数通过加大前景区域的权重来避免网络陷入损失函数的局部最小值,即避免发生过拟合现象。因此,Dice损失函数较适用于样本极度不平衡的情况。
根据式(4)可求得Dice损失函数关于
| $ \frac{2\times {{G}_{\mathrm{G}\mathrm{T}}}^{2}}{({G}_{\mathrm{G}\mathrm{T}}+{P}_{\mathrm{P}\mathrm{R}}{)}^{2}} $ | (5) |
当
基于以上分析可知,BCEWithLogits和Dice损失函数都有优点和局限性,因此,BCEWithLogits和Dice相结合的损失函数如式(6)所示:
| $ {L}_{\mathrm{总}}=\lambda \times {L}_{\mathrm{B}\mathrm{C}\mathrm{E}\mathrm{W}\mathrm{i}\mathrm{t}\mathrm{h}\mathrm{L}\mathrm{o}\mathrm{g}\mathrm{i}\mathrm{t}\mathrm{s}}+(1-\lambda )\times {L}_{\mathrm{D}\mathrm{i}\mathrm{c}\mathrm{e}} $ | (6) |
其中:
为验证Concat-UNet网络对食管癌肿瘤进行快速精准分割的有效性,本文在真实CT影像数据集上进行实验,并与U-Net、SegNet、U-Net_LI、ERFNet网络在同样的实验环境下进行对比。
3.1 数据集预处理数据集是从医院获取的真实CT影像,包含原始数据以及医生已标注的数据,数据格式为DICOM,切片大小为512×512像素。为了便于数据读取及网络训练,本文需对数据集进行预处理。
食管癌肿瘤大致位于胸部CT影像的中间部分且所占比例很小,如果直接使用原始大小的CT影像进行训练,网络易受类别不平衡的干扰,影响模型的训练和最终的分割效果。因此,本文根据食管癌肿瘤的位置统一将图片裁剪为256×256像素,制作标准的数据标签,数据标签同样为256×256像素的二值化png格式的图片,处理后的图片只包含食管癌肿瘤区域和背景区域,其中,肿瘤区域的值为1,背景区域的值为0。最后,将数据集划分为两部分,一部分用于网络的训练,另一部分用于网络的测试,在训练时按照7∶3的比例随机分为训练集和验证集。
3.2 实验环境本文实验使用深度学习框架PyTorch在云环境中进行训练,云环境使用Google提供的Colaboratory。在训练过程中为了能够在模型收敛的情况下得到最优的模型,同时在数据量较少的情况下降低过拟合现象的发生。在实验过程中引入早停机制,最终的迭代次数不是人为设定的而是由验证集上的损失值决定,最大迭代次数设置为400。受数据集以及实验环境的限制,训练过程中的batch size大小设定为6。本文选择自适应参数的优化算法RMSProp,学习率初始化为0.05,早停机制中的耐心值为40。
3.3 评价指标医学图像分割领域有很多评价指标,它们能够从不同的角度对网络的预测结果进行评估。针对食管癌肿瘤的分割,本文将肿瘤区域视为正样本,将非肿瘤区域视为负样本,根据样例真实类别与预测类别的组合划分为真正例(True Positive,
本文采用精确率(P)、召回率(R)、F1-Score、Dice系数(Dice)作为评价指标。
1)精确率(P),计算所有预测结果为正(正样本被预测为正
| $ P=\frac{{T}_{\mathrm{T}\mathrm{P}}}{{T}_{\mathrm{T}\mathrm{P}}+{F}_{\mathrm{F}\mathrm{P}}} $ | (7) |
在肿瘤分割问题中,精确率表征预测的肿瘤区域中真正肿瘤所占的比例。
2)召回率(R),计算所有预测结果正确(正样本被预测为正
| $ R=\frac{{T}_{\mathrm{T}\mathrm{P}}}{{T}_{\mathrm{T}\mathrm{P}}+{F}_{\mathrm{F}\mathrm{N}}} $ | (8) |
在肿瘤分割问题中,召回率表征所有肿瘤区域中预测成功的样本所占比例。
3)F1-Score,是精确率和召回率的调和平均数,如式(9)所示:
| $ \mathrm{F}1 - \mathrm{S}\mathrm{c}\mathrm{o}\mathrm{r}\mathrm{e}=\frac{2\times (P\times R)}{P+R} $ | (9) |
精确率主要是预测肿瘤区域中预测正确的概率,它是基于预测的肿瘤区域进行评估,而忽略了真正的肿瘤区域。召回率只关注真正肿瘤区域中有多少样本被正确预测出来,它是基于真正的肿瘤区域进行评估,忽略了预测区域中预测错误的样本。因此,精确率和召回率的评估都不够全面。在对肿瘤图像预测时,精确率和召回率同等重要,F1-Score既考虑了预测的肿瘤区域,又考虑了真正的肿瘤区域,评估结果比较全面。
4)Dice系数,将精确率和召回率相结合的评估指标。根据式(3)可知,Dice系数既考虑了预测的肿瘤区域,又考虑了真正的肿瘤区域,评估结果比较全面,在实验过程中将Dice系数作为网络评估指标。
3.4 结果分析食管癌肿瘤定位的关键是能够对肿瘤区域进行精确的分割。为验证Concat-UNet网络的分割效果及肿瘤区域的定位情况,本文与SegNet、ERFNet、U-Net以及文献[25]提出的U-Net_LI网络进行对比,评估基准网络U-Net和Concat-UNet的网络性能以及其他网络的Dice系数、精确率、召回率以及F1-Score。
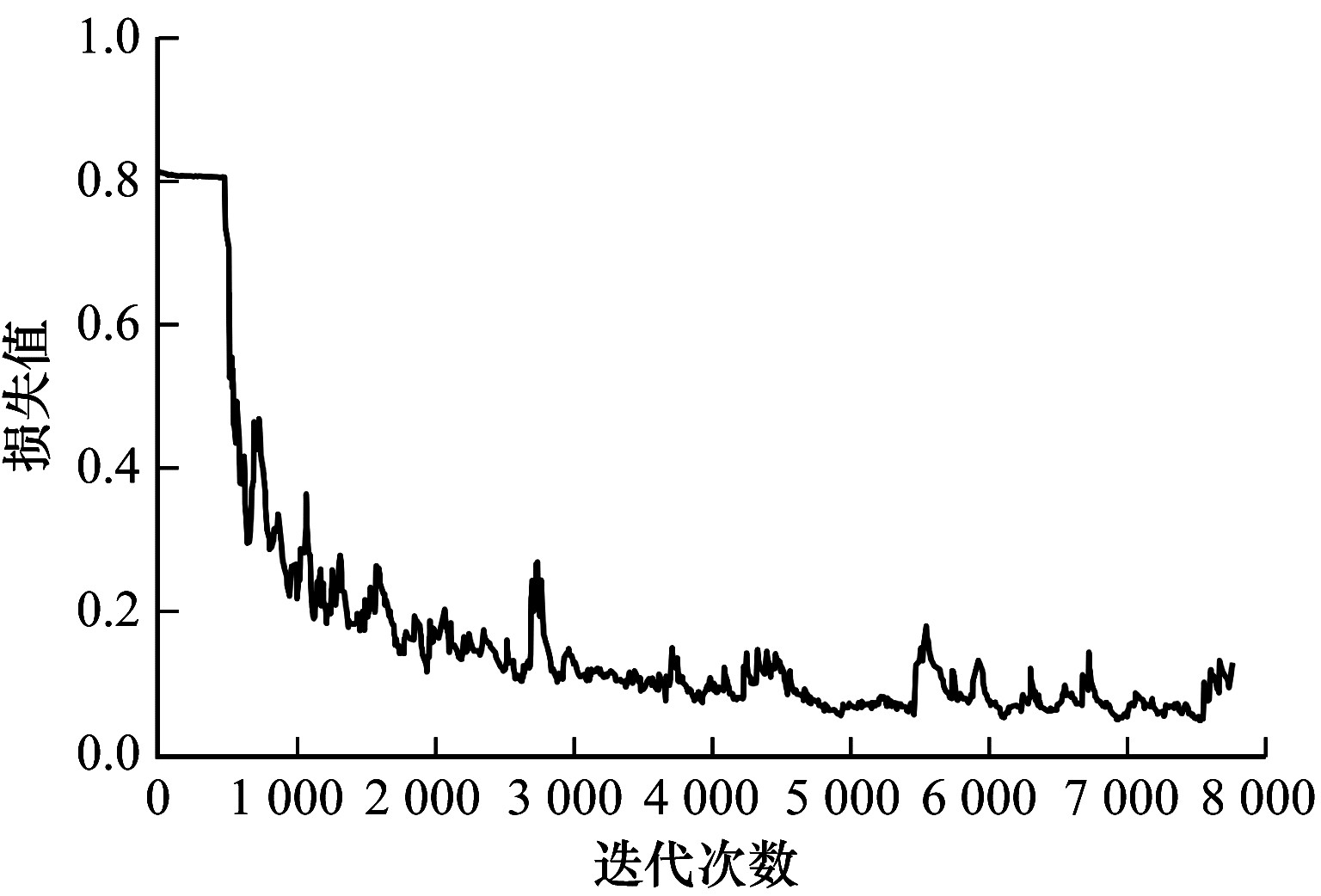
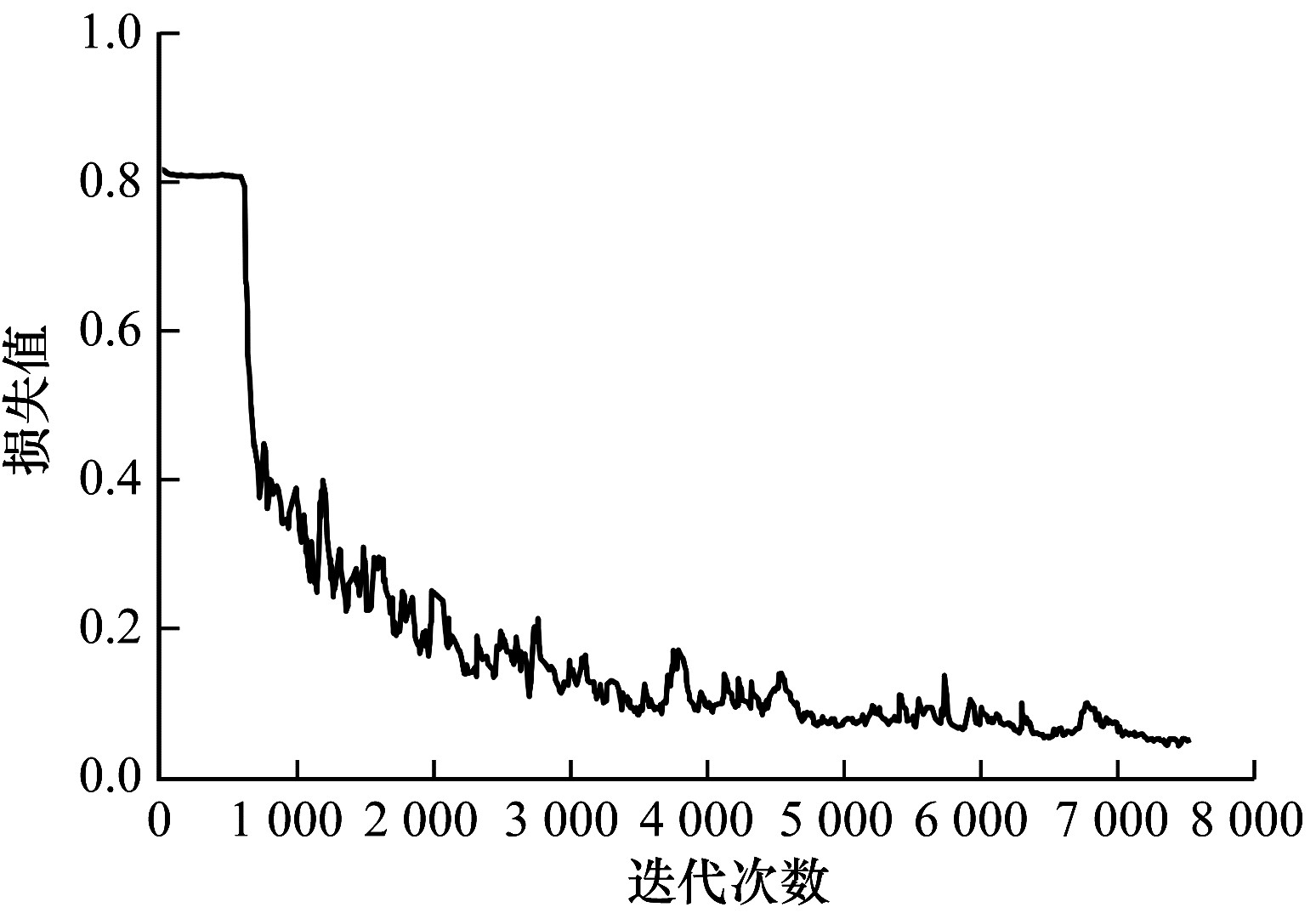
3.4.1 网络性能对比为了更直观地对比U-Net网络与Concat-UNet网络训练时的性能,本文分别记录这2个网络在训练过程中训练集上的损失值和验证集上的得分情况。随着训练迭代次数的增大,U-Net和Concat-UNet网络训练过程中损失值的对比如图 5和图 6所示。
|
Download:
|
| 图 5 U-Net网络的损失值 Fig. 5 Loss values of the U-Net network | |
|
Download:
|
| 图 6 Concat-UNet网络的损失值 Fig. 6 Loss values of the Concat-UNet network | |
从图 5和图 6可以看出,相比U-Net,Concat-UNet网络在训练过程中曲线下降趋势更加平稳,说明Concat-UNet更加稳定,模型更容易收敛,具有较优的数据泛化性能。
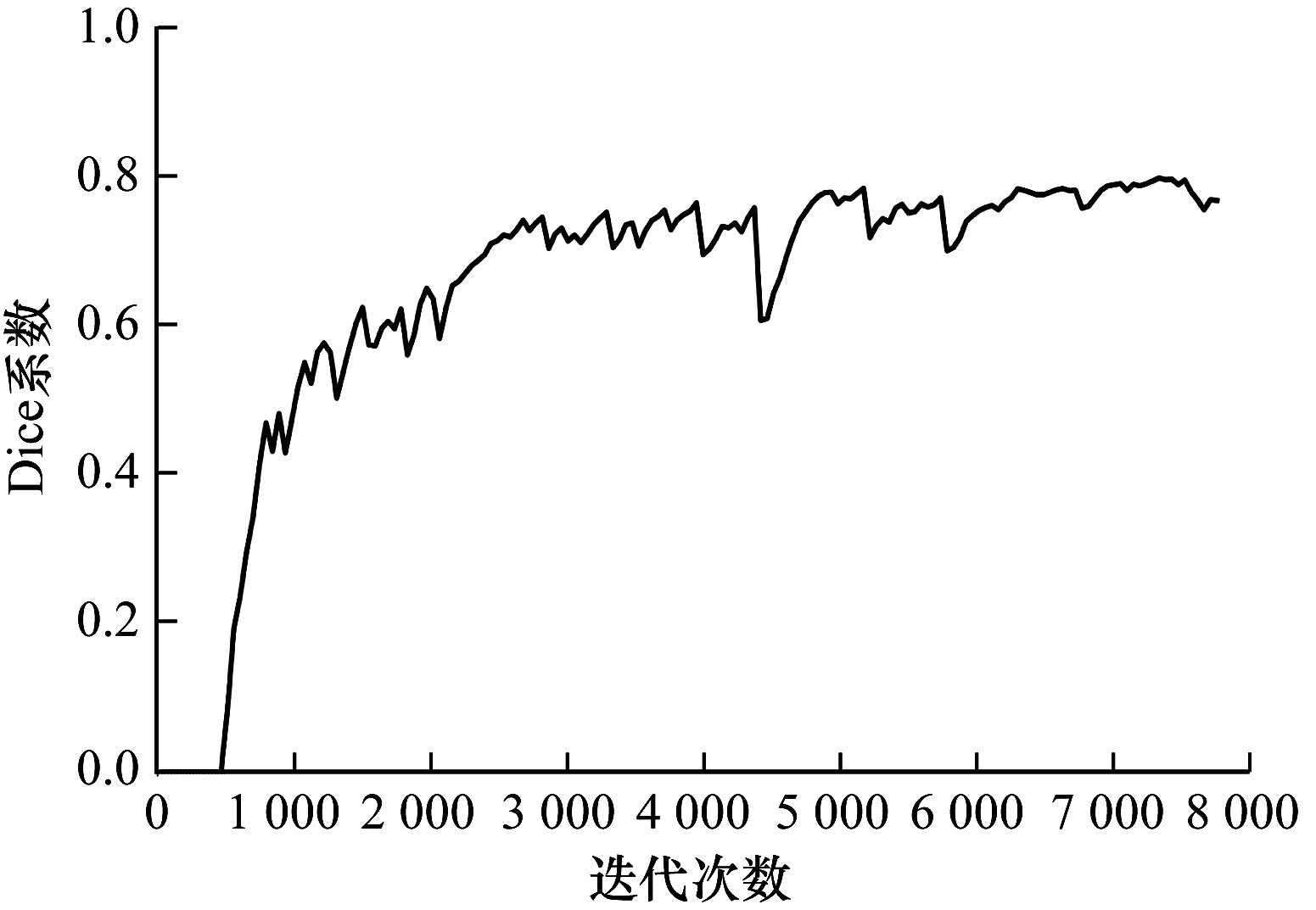
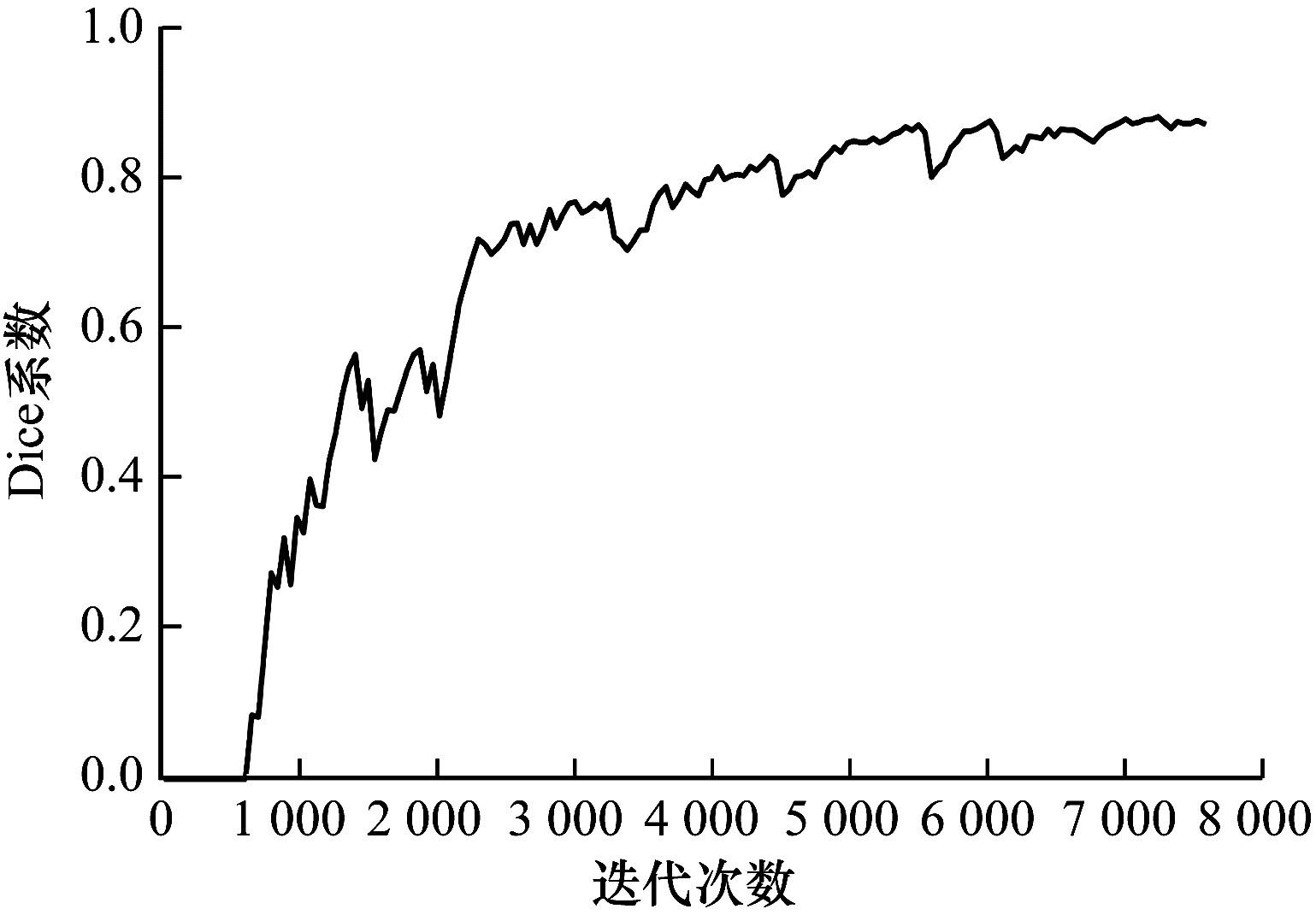
随着测试迭代次数的增大,U-Net和Concat-UNet网络训练过程中Dice系数的对比如图 7和图 8所示。从图 7和图 8可以看出,Concat-UNet的Dice系数曲线较U-Net的Dice系数曲线更平稳。此外,在训练趋于稳定时对应验证集上的得分较U-Net也有明显的提高,即网络的分割效果有所提升。Concat-UNet通过在Conv_concat模块中引入2个跳跃连接,使数据在经过网络卷积模块后丢失更少的有用信息,有利于图像特征的提取,在一定程度上提高了分割精度,同时在卷积模块后引入的BN层也使网络训练更加稳定。
|
Download:
|
| 图 7 U-Net网络的Dice系数变化曲线 Fig. 7 Dice coefficient change curve of the U-Net network | |
|
Download:
|
| 图 8 Concat-UNet网络的Dice系数变化曲线 Fig. 8 Dice coefficient change curve of the Concat-UNet network | |
从测试集中选取5张不同的CT影像,SegNet、ERFNet、U-Net、U-Net_LI、Concat-UNet网络对影像中的肿瘤特征进行提取,提取结果如图 9所示。图 9(a)是原始的CT影像经过处理后得到的训练图像,图 9(b)中的白色位置为肿瘤。从图 9可以看出,SegNet对肿瘤特征的识别效果较差,这是由于SegNet在解码器上采样过程中的输入直接使用了编码器对应位置的最大池化层输出,没有为网络学习一个新的参数层,而对于食管癌的CT图像,肿瘤区域与周围区域的边界并不明显,这是造成SegNet分割效果较差的原因。ERFNet提取效果相对原始的肿瘤区域形状不太贴合,检测的肿瘤区域边缘不平滑且出现少许锯齿状边缘,并且对肿瘤的边缘噪声更加敏感。U-Net提取的肿瘤区域边缘较为平滑,相对于原始肿瘤区域更大,在第3列的示例中还出现了误检的情况,表示U-Net对数据的泛化性较差。U-Net_LI网络主要解决图像分割中的轮廓图小部分不连续问题,能更好地处理肿瘤区域的边缘,使得分割的肿瘤边缘更平滑,但也会损失细节信息。Concat-UNet网络提取的肿瘤区域与原始肿瘤区域在形状和大小上更为贴合,并且在第3列的示例中相对于U-Net不会出现误检的情况。因此,Concat-UNet在检测精确度和准确度方面要优于其他网络。

|
Download:
|
| 图 9 不同网络的肿瘤分割结果对比 Fig. 9 Segmentation results comparison among different networks | |
本文在220个样例的数据集上对5种网络均重复训练5次,根据同一测试集上的测试结果保存最优的网络参数,然后分别用最优的SegNet、ERFNet、U-Net、U-Net_LI、Concat-UNet网络对同一个测试集进行肿瘤分割,并计算5种网络在评价指标Dice、P、R以及F1-Score上的均值,评估结果如表 1所示。从表 1可以看出,SegNet召回率最低,表示所有肿瘤区域中预测成功的样本比例较低。ERFNet相较于SegNet具有较优的结果,能较好地权衡精确率与召回率,但是各项指标都低于Concat-UNet。U-Net网络的召回率与精确率相差很大,召回率很高,但精确率很低,这表明U-Net网络的肿瘤提取不够准确。U-Net_LI在精确率上较U-Net有明显提升,但效果仍不及Concat-UNet。Concat-UNet网络在保证召回率适中的情况下相比U-Net网络精确度提升了11.64个百分点,实现了精确率与召回率的平衡。因此,Concat-UNet网络在综合评估指标Dice和F1-Score上也有明显的提升。综合4种评估结果,Concat-UNet网络的整体表现优于其他网络。
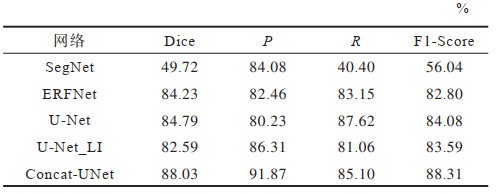
|
下载CSV 表 1 不同网络的评估指标对比 Table 1 Evaluation indexs comparison among different networks |
为了更直观地看出食管癌肿瘤在CT影像上的位置,本文对Concat-UNet网络预测后的二值化图片进行处理,通过轮廓提取与标注完成CT影像上食管癌肿瘤的分割任务。Concat-UNet网络的肿瘤分割可视化结果如图 10所示。从图 10可以看出,Concat-UNet网络能够实现对肿瘤区域的精准分割。

|
Download:
|
| 图 10 Concat-UNet肿瘤分割的可视化结果 Fig. 10 Visualization results of Concat-UNet tumor segmentation | |
本文采用的损失函数
在Concat-UNet网络上分别进行4组实验,将每组实验中Dice损失函数所占的比例逐渐增大,在同一个数据集上进行训练,再对同一个测试集进行评估,评估结果如表 2所示。当
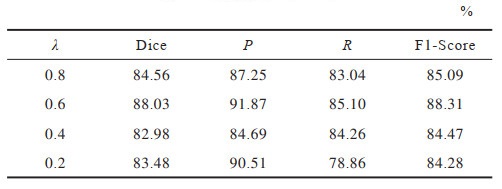
|
下载CSV 表 2 不同比例的Dice损失函数对Concat-UNet网络评估结果的影响 Table 2 Influence of different proportions of Dice loss function on evaluation results of Concat-UNet network |
本文提出图像分割网络Concat-UNet。在卷积块中引入跳跃连接和批量归一化层,并改进损失函数,以提高特征学习的效率和网络的优化效果。实验结果表明,相比传统U-Net网络,Concat-UNet能够精准地对影像中的食管癌肿瘤进行分割标注,为今后食管癌肿瘤的诊断工作提供一种新的解决方法。下一步将CT影像切片的2D图像改进为3D影像,结合CT影像中的3D空间信息识别并提取肿瘤区域。此外,将本文工作应用到实际的医学影像系统中,构建CT影像拍片、标注、诊断一体式智能医学影像系统也是下一步的重点研究方向。
| [1] |
ARNOLD M, SOERJOMATARAM I, FERLAY J, et al. Global incidence of oesophageal cancer by histological subtype in 2012[J]. Gut, 2015, 64(3): 381-387. DOI:10.1136/gutjnl-2014-308124 |
| [2] |
CHEN W Q, ZHENG R S, BAADE P D, et al. Cancer statistics in China, 2015[J]. CA: A Cancer Journal for Clinicians, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 |
| [3] |
李鹏, 王拥军, 陈光勇, 等. 中国巴雷特食管及其早期腺癌筛查与诊治共识(2017万宁)[J]. 中国实用内科杂志, 2017, 37(9): 798-809. LI P, WANG Y J, CHEN G Y, et al. Chinese consensus: screening, diagnosis and management of Barrett's esophagus and adenocarcinoma(2017 Wanning)[J]. Chinese Journal of Medicine, 2017, 37(9): 798-809. (in Chinese) |
| [4] |
丘文峰. 基于Python的医学图像处理框架及其应用[D]. 广州: 华南师范大学, 2010. QIU W F. Python-based medical image processing framework and its application [D]. Guangzhou: South China Normal University, 2010. (in Chinese) |
| [5] |
俞益洲, 马杰超, 石德君, 等. 深度学习在医学影像分析中的应用综述[J]. 数据与计算发展前沿, 2019, 1(6): 37-52. YU Y Z, MA J C, SHI D J, et al. Application of deep learning in medical imaging analysis: a survey[J]. Frontiers of Data & Computing, 2019, 1(6): 37-52. (in Chinese) |
| [6] |
王弈, 李传富. 人工智能方法在医学图像处理中的研究新进展[J]. 中国医学物理学杂志, 2013, 30(3): 4138-4143. WANG Y, LI C F. The new research progress of artificial intelligent methods in medical image processing[J]. Chinese Journal of Medical Physics, 2013, 30(3): 4138-4143. (in Chinese) DOI:10.3969/j.issn.1005-202X.2013.03.013 |
| [7] |
IOFFE S, SZEGEDY C. Batch normalization: accelerating deep network training by reducing internal covariate shift[C]//Proceedings of the 32nd International Conference on Machine Learning. New York, USA: ACM Press, 2015: 448-456.
|
| [8] |
MUTHUKRISHNAN R, RADHA M. Edge detection techniques for image segmentation[J]. International Journal of Computer Science & Information Technology, 2012, 3(6): 250-254. |
| [9] |
KAGANAMI H G, ZOU B J. Region-based segmentation versus edge detection[C]//Proceedings of the 5th International Conference on Intelligent Information Hiding and Multimedia Signal Processing. Washington D. C., USA: IEEE Press, 2009: 1217-1221.
|
| [10] |
SHI J B, MALIK J. Normalized cuts and image segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2000, 22(8): 888-905. DOI:10.1109/34.868688 |
| [11] |
BOYKOV Y, FUNKA-LEA G. Graph cuts and efficient N-D image segmentation[J]. International Journal of Computer Vision, 2006, 70(2): 109-131. DOI:10.1007/s11263-006-7934-5 |
| [12] |
ROTHER C. "GrabCut": Interactive foreground extraction using iterated graph cuts[J]. Proceedings of Siggraph, 2004, 23(3): 309-314. DOI:10.1145/1015706.1015720 |
| [13] |
COATES A, ANDREW Y. Learning feature representations with K-means[EB/OL]. [2021-08-01]. https://citeseerx.ist.psu.edu/viewdoc/download;jsessionid=17E81A9ECB385E299D5CC6021E8F3871?doi=10.1.1.269.5607&rep=rep1&type=pdf.
|
| [14] |
LUXBURG U. A tutorial on spectral clustering[J]. Statistics and Computing, 2007, 17(4): 395-416. DOI:10.1007/s11222-007-9033-z |
| [15] |
COMANICIU D, RAMESH V, MEER P. Real-time tracking of non-rigid objects using mean shift[C]//Proceedings of Conference on Computer Vision and Pattern Recognition. Washington D. C., USA: IEEE Press, 2003: 142-149.
|
| [16] |
ACHANTA R, SHAJI A, SMITH K, et al. SLIC superpixels compared to state-of-the-art superpixel methods[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2012, 34(12): 2274-2282. DOI:10.1109/TPAMI.2012.120 |
| [17] |
吴永芳, 杨鑫, 徐敏, 等. 基于K均值聚类的图割医学图像分割算法[J]. 计算机工程, 2011, 37(5): 232-234. WU Y F, YANG X, XU M, et al. Graph cuts medical image segmentation algorithm based on K-means clustering[J]. Computer Engineering, 2011, 37(5): 232-234. (in Chinese) |
| [18] |
杨柳, 陈永林, 王翊, 等. 基于核图割模型的肝脏CT图像肿瘤分割[J]. 计算机工程, 2014, 40(3): 238-243. YANG L, CHEN Y L, WANG Y, et al. Tumor segmentation for liver CT images based on kernel graph cut model[J]. Computer Engineering, 2014, 40(3): 238-243. (in Chinese) DOI:10.3969/j.issn.1000-3428.2014.03.050 |
| [19] |
SHELHAMER E, LONG J, DARRELL T. Fully convolutional networks for semantic segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(4): 640-651. DOI:10.1109/TPAMI.2016.2572683 |
| [20] |
TONG N, GOU S P, YANG S Y, et al. Fully automatic multi-organ segmentation for head and neck cancer radiotherapy using shape representation model constrained fully convolutional neural networks[J]. Medical Physics, 2018, 45(10): 4558-4567. DOI:10.1002/mp.13147 |
| [21] |
RONNEBERGER O, FISCHER P, BROX T. U-Net: convolutional networks for biomedical image segmentation[C]//Proceedings of International Conference on Medical Image Computing and Computer-Assisted Intervention. Berlin, Germany: Springer, 2015: 234-241.
|
| [22] |
DROZDZAL M, VORONTSOV E, CHARTRAND G, et al. The importance of skip connections in biomedical image segmentation[C]//Proceedings of Deep Learning and Data Labeling for Medical Applications. New York, USA: Springer, 2016: 179-187.
|
| [23] |
BROSCH T, TANG L Y W, YOO Y, et al. Deep 3D convolutional encoder networks with shortcuts for multiscale feature integration applied to multiple sclerosis lesion segmentation[J]. IEEE Transactions on Medical Imaging, 2016, 35(5): 1229-1239. DOI:10.1109/TMI.2016.2528821 |
| [24] |
林志洁, 郑秋岚, 梁涌, 等. 基于内卷U-Net的医学图像分割模型[J]. 计算机工程, 2022, 48(8): 180-186. LIN Z J, ZHENG Q L, LIANG Y, et al. Medical image segmentation model based on involution U-Net[J]. Computer Engineering, 2022, 48(8): 180-186. (in Chinese) |
| [25] |
仵晨阳, 何瑶. 基于U-Net网络的食管癌病灶的分割研究[J]. 计算机与数字工程, 2020, 48(12): 2734-2738. WU C Y, HE Y. Segmentation of esophageal cancer lesions based on U-Net network[J]. Computer & Digital Engineering, 2020, 48(12): 2734-2738. (in Chinese) DOI:10.3969/j.issn.1672-9722.2020.11.038 |
| [26] |
KRÄHENBÜHL P, KOLTUN V. Efficient inference in fully connected CRFs with Gaussian edge potentials[C]//Proceedings of the 24th International Conference on Neural Information Processing Systems. New York, USA: ACM Press, 2011: 109-117.
|
| [27] |
CHEN L C, PAPANDREOU G, KOKKINOS I, et al. DeepLab: semantic image segmentation with deep convolutional nets, atrous convolution, and fully connected CRFs[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2018, 40(4): 834-848. DOI:10.1109/TPAMI.2017.2699184 |
| [28] |
CHEN L C, PAPANDREOU G, SCHROFF F, et al. Rethinking atrous convolution for semantic image segmentation[EB/OL]. [2021-08-01]. https://arxiv.org/pdf/1706.05587.pdf.
|
| [29] |
LIU W, RABINOVICH A, BERG A C. ParseNet: looking wider to see better[EB/OL]. [2021-08-01]. https://arxiv.org/pdf/1506.04579.pdf.
|
| [30] |
BADRINARAYANAN V, KENDALL A, CIPOLLA R. SegNet: a deep convolutional encoder-decoder architecture for image segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(12): 2481-2495. DOI:10.1109/TPAMI.2016.2644615 |
| [31] |
ROMERA E, ÁLVAREZ J M, BERGASA L M, et al. ERFNet: efficient residual factorized ConvNet for real-time semantic segmentation[J]. IEEE Transactions on Intelligent Transportation Systems, 2018, 19(1): 263-272. DOI:10.1109/TITS.2017.2750080 |
| [32] |
HE K M, ZHANG X Y, REN S Q, et al. Deep residual learning for image recognition[C]//Proceedings of Conference on Computer Vision and Pattern Recognition. Washington D. C., USA: IEEE Press, 2016: 770-778.
|
| [33] |
GLOROT X, BORDES A, BENGIO Y. Deep sparse rectifier neural networks[C]//Proceedings of the 14th International Conference on Artificial Intelligence and statistics. New York, USA: ACM Press, 2011: 315-323.
|
 2022, Vol. 48
2022, Vol. 48

 ,
,